R資料分析:整合學習方法之隨機生存森林的原理和做法,範例解析
很久很久以前給大家寫過決策樹,非常簡單明瞭的演演算法。今天給大家寫隨機(生存)森林,隨機森林是整合了很多個決策數的整合模型。像隨機森林這樣將很多個基本學習器集合起來形成一個更加強大的學習器的這麼一種整合思想還是非常好的。所以今天來寫寫這類演演算法。

整合學習方法
Ensemble learning methods are made up of a set of classifiers—e.g. decision trees—and their predictions are aggregated to identify the most popular result.
所謂的整合學習方法,就是把很多的比較簡單的學習演演算法統起來用,比如光看一個決策樹,好像效果比較單調,還比較容易過擬合,我就訓練好多樹,把這些樹的結果綜合一下,結果應該會好很多,用這麼樣思路形成的演演算法就是整合學習演演算法Ensemble methods,就是利用很多個基礎學習器形成一個綜合學習器。
Basically, a forest is an example of an ensemble, which is a special type of machine learning method that averages simple functions called base learners.The resulting averaged learner is called the ensemble
整合學習方法最有名的就是bagging 和boosting 方法:
The most well-known ensemble methods are bagging, also known as bootstrap aggregation, and boosting
BAGGing
BAGGing, or Bootstrap AGGregating這個方法把自助抽樣和結果合併整合在一起,包括兩個步驟,一個就是自助抽樣,抽很多個資料集出來,每個資料集來訓練一個模型,這樣就可以有很多個模型了;第二步就是將這麼多模型的結果合併出來最終結果,這個最終結果相對於單個模型結果就會更加穩健。
In the bagging algorithm, the first step involves creating multiple models. These models are generated using the same algorithm with random sub-samples of the dataset which are drawn from the original dataset randomly with bootstrap sampling method
The second step in bagging is aggregating the generated models.
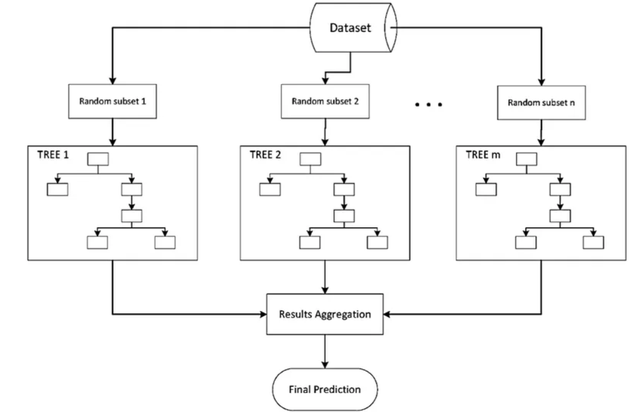
隨機森林就可以看作是遵循了bagging方法的一個思路,只不過在每一個抽樣樣本中的樹(模型)是不一樣的:
Boosting:
Boosting為強化學習,最大的特點是可以將原來的弱模型變強,邏輯在於演演算法會先後訓練很多模型,後面訓練模型的時候會不斷地給原來模型表現不好的樣本增大權重,使得後面的模型越來越將學習重點放在之前模型表現差的樣本上,這麼一來,整體模型越來越強。就像人會從之前的錯誤中反省經驗一個意思了。
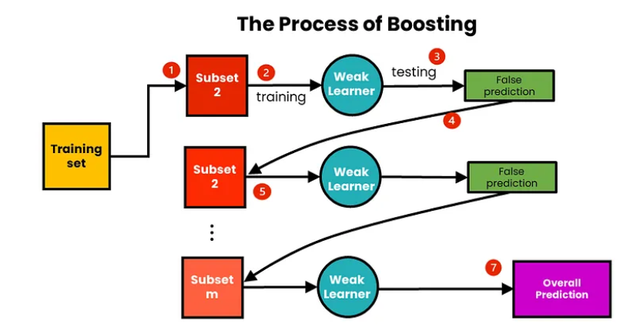
這麼一描述大家就知道,boosting方法的模型訓練是有先後順序的,並行演演算法就用不了了
Boosting incrementally builds an ensemble by training each model with the same dataset but where the weights of instances are adjusted according to the error of the last prediction.
Boosting方法本身也有很多,常見的如AdaBoost,Gradient Boosting(XGBoost and LightGBM),下圖感興趣的同學可以看看:
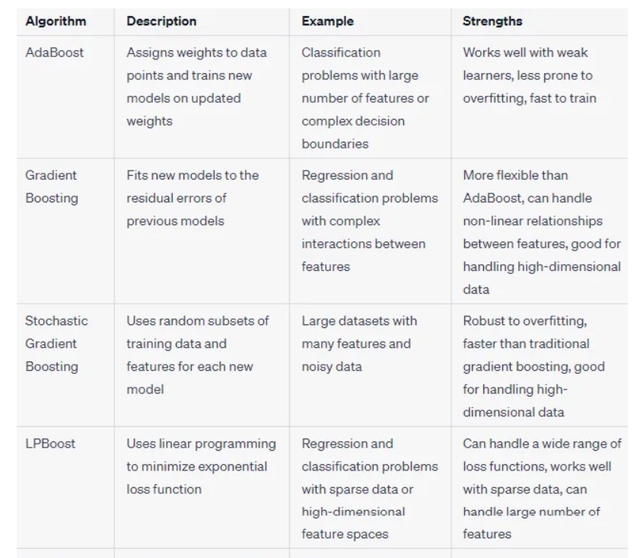
上面的演演算法之後再給大家寫,接下來的實操部分還是以隨機森林為例子給大傢俱體介紹:
隨機森林
隨機森林模型的擬合過程大概可以分為三步:
1.通過有放回的自助抽樣形成ntree個抽樣樣本集(Bootstrap)
2.對每個抽樣樣本集形成一個決策樹,這個樹是基於mtry個預測因子的
3.將最終的模型結果就是ntree個抽樣樣本集得出的結果的最大票數或者均值(AGGregating)
隨機森林的整個的流程就如下圖:
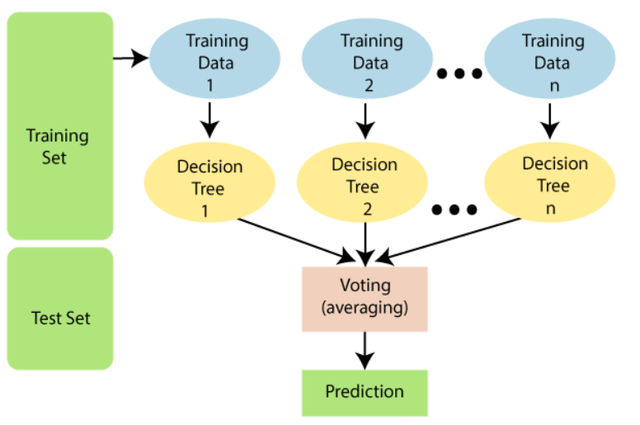
為了方便理解「最終的模型結果就是ntree個抽樣樣本集得出的結果的最大票數或者均值」我們用例子做個解釋,先看下圖:
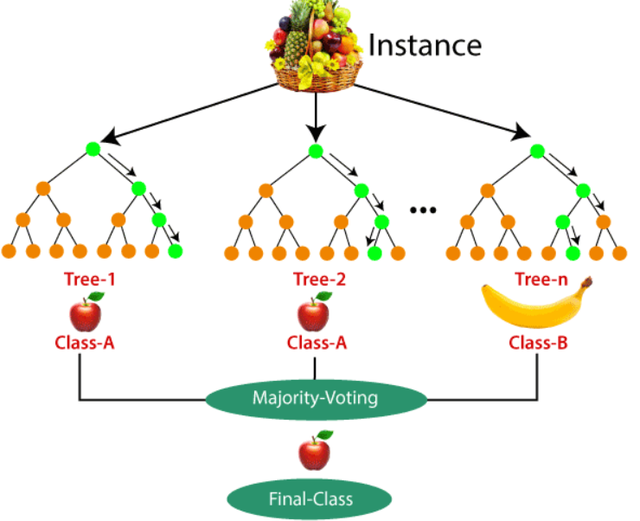
我們有一個水果集,然後我訓練一個3棵樹組成的隨機森林來判斷每一個水果到底是何種類,有兩棵樹都告訴我是某一個水果是蘋果,一棵樹告訴我是香蕉,那麼最後我們隨機森林就會輸出該水果是香蕉的結論。
上面的過程有幾個超參需要確定
- mtry: Number of variables randomly sampled as candidates at each split.
- ntree: Number of trees to grow.
mtry一般需要調參,ntree都是越大越好自己設定就行。在上面的過程中我們每棵樹的節點都是不同的,叫做特徵隨機化,通過特徵隨機化我們保證了森林中樹的多樣性,隨機森林模型也更加穩健。
Feature randomness, also known as feature bagging or 「the random subspace method」, generates a random subset of features, which ensures low correlation among decision trees
隨機森林實操
比如我現在有一個資料集,結局變數是class為二分類,我要適用隨機森林演演算法就可以寫出如下程式碼:
rf_default <- train(Class~.,
data=dataset,
method='rf',
tuneLength = 15,
trControl=control)
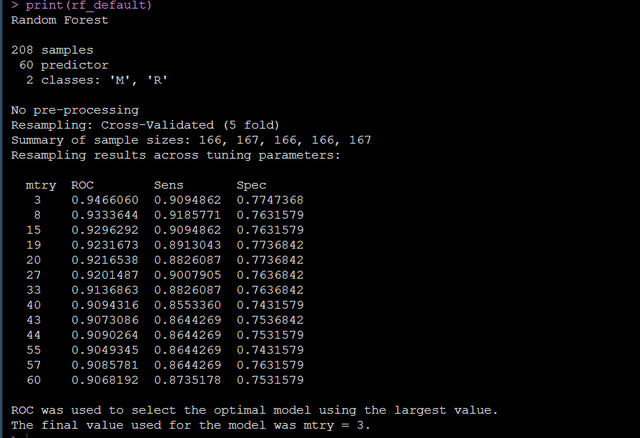
print(rf_default)輸出的結果中有隨機調參的過程,共15次,最終發現超參mtry=3的時候模型最優,具體如下:
以上的隨機森林模型的簡單展示,接著我們再看隨機生存森林。
隨機生存森林
和隨機森林一樣,隨機生存森林也是一個整合學習方法,區別在於其結局為生存資料。
範例文章
依然我們來看一篇發表在Cancer Med.上的文章,名字如下:
Prognostic risk factor of major salivary gland carcinomas and survival prediction model based on random survival forests
作者用cox進行了變數篩選,使用隨機生存森林進行了預測模型構建,並得到了相應的風險分,明確了風險分的最佳截斷值(「maxstat」 R package),對於模型的表現作者使用了c指數和time-dependent ROC來評估,文章中主要的結果報告如下,包括:
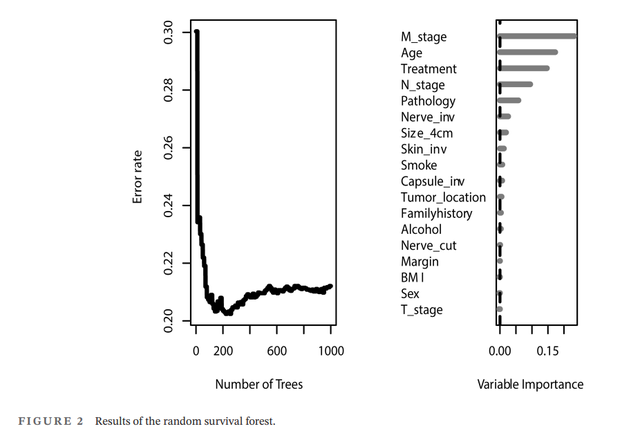
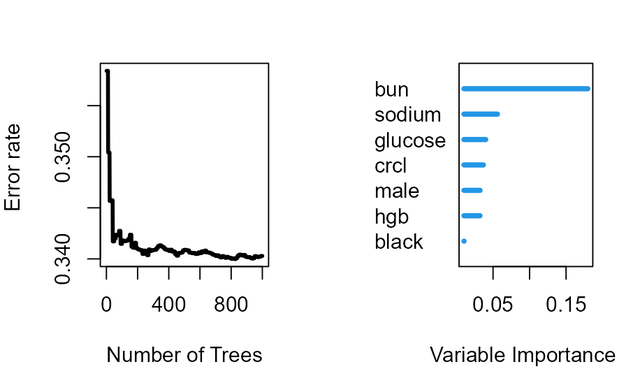
樹的數量和模型誤差情況,以及變數重要性的結果:
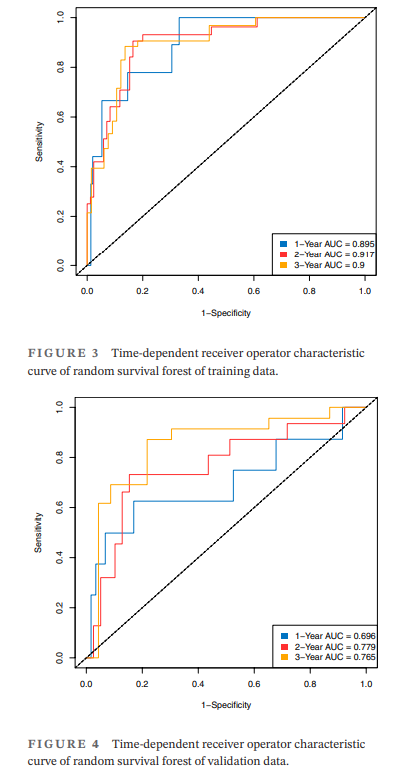
time-dependent ROC曲線結果展示和相應的AUC值:
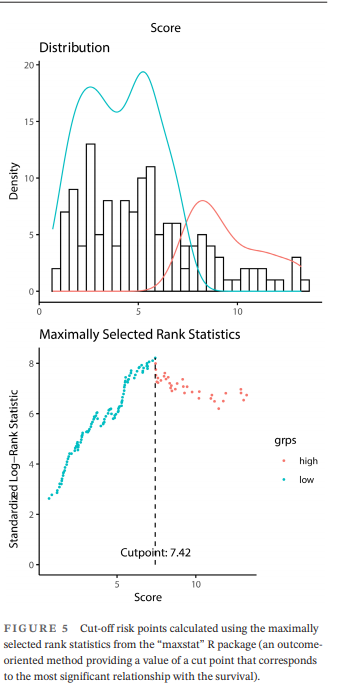
風險分界址點確定:
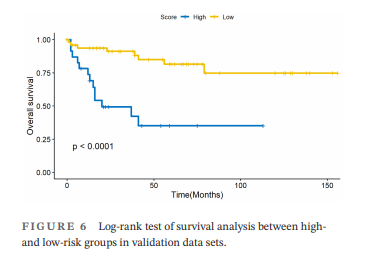
高低風險組的組間生存曲線比較:
也是一篇預測模型類文章的常規套路了。挑一個演演算法,擬合模型後評估,做個風險分,應用風險分劃分病人證明模型可用性。我們以這篇文章為例子看隨機生存森林預測模型的實操。
隨機生存森林範例操作
我現在的資料中ttodead,died兩個變數分別是時間和生存狀態,此時我想做一個隨機生存森林模型就可以寫出如下程式碼:
RF_obj <- rfsrc(Surv(ttodead,died)~., dataSet, ntree = 1000, membership = TRUE, importance=TRUE)對程式碼執行後生成的物件RF_obj進行plot即可出圖如下,就得到了原文中的figure2:
然後我們可以畫出模型的不同時間點的timeRoc曲線(下面程式碼中的risk_score為隨機生存森林物件的預測值),就得到了原文中的figure3,figure4:
ROC_rsf<-timeROC(T=finaldata.Test$Surv_day,delta=finaldata.Test$status,
marker=risk_score,
cause=1,
times=c(365,365*3,365*5),iid=TRUE)
plot(ROC_lasso,time=365)
plot(ROC_lasso,time=365*3,add = T,col="blue")
plot(ROC_lasso,time=365*5,add = T,col="green")
legend(.8, .3, legend=c("T=1 Year AUC=0.895", "T=3 Year AUC=0.917","T=5 Year AUC=0.926"),
col=c("red", "blue","green"), lty=1, cex=0.7,bty = "n")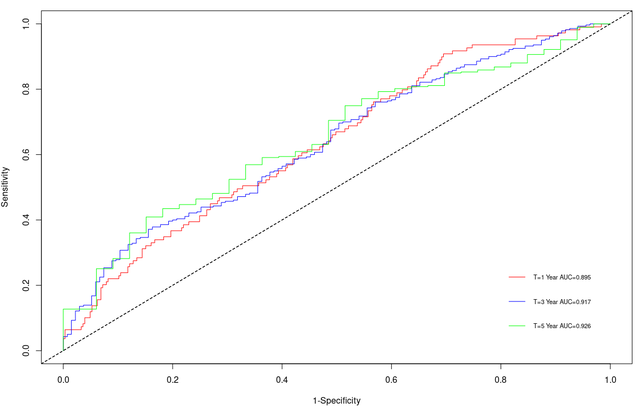
並且將模型預測值的截斷值找出來,驗證模型在不同風險組的區分能力。其中找風險分截斷值的程式碼如下:
y.pred <- predict(RF_obj)[["predicted"]]
plot(surv_cutpoint(dataSet, time = "ttodead", event = "died",
variables = c("y.pred")), "y.pred", palette = "npg")執行後得到下圖(原文中的figure5),就說明我們這個模型的風險分截斷值應該為43.21:
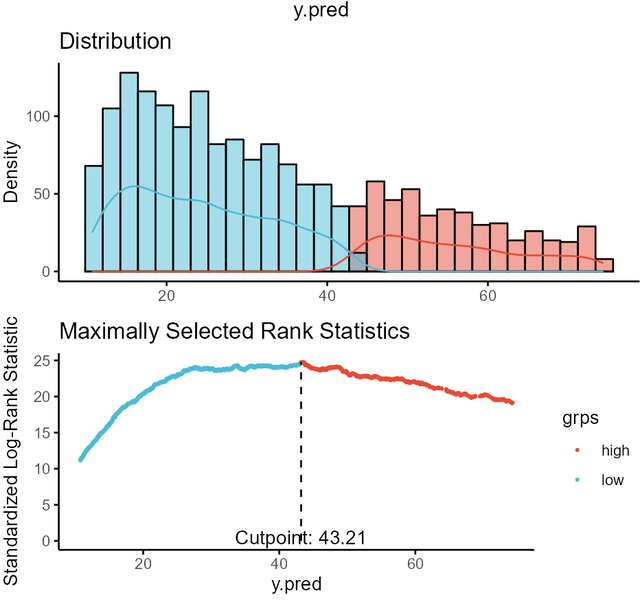
然後根據這個風險分我們就可以將原始人群分為高風險組和低風險組,再做出組間km曲線,到這兒相當於Cancer Med的這篇用隨機生存森林的文章就完全復現出來了。
以上是給大家介紹的隨機生存森林的內容。