【scipy 基礎】--聚類
物以類聚,聚類演演算法使用最佳化的演演算法來計算資料點之間的距離,並將它們分組到最近的簇中。
Scipy的聚類模組中,進一步分為兩個聚類子模組:
vq(vector quantization):提供了一種基於向量量化的聚類演演算法。
vq模組支援多種向量量化演演算法,包括K-means、GMM(高斯混合模型)和WAVG(均勻分佈)。
hierarchy:提供了一種基於層次聚類的聚類演演算法。
hierarchy模組支援多種層次聚類演演算法,包括ward、elbow和centroid。
總之,Scipy中的vq和hierarchy模組都提供了一種基於最小化平方誤差的聚類演演算法,
它們可以幫助我們快速地對大型資料集進行分組,從而更好地理解資料的分佈和模式。
1. vq 聚類
vq 聚類演演算法的原理是將資料點對映到一組稱為「超空間」的低維向量空間中,然後將它們分組到最近的簇中。
首先,我們建立一些測試資料:(建立3個類別的測試資料)
import numpy as np
import matplotlib.pyplot as plt
data1 = np.random.randint(0, 30, (100, 3))
data2 = np.random.randint(30, 60, (100, 3))
data3 = np.random.randint(60, 100, (100, 3))
data = np.concatenate([data1, data2, data3])
fig, ax = plt.subplots(subplot_kw={"projection": "3d"})
ax.scatter(data[:, 0], data[:, 1], data[:, 2])
plt.show()
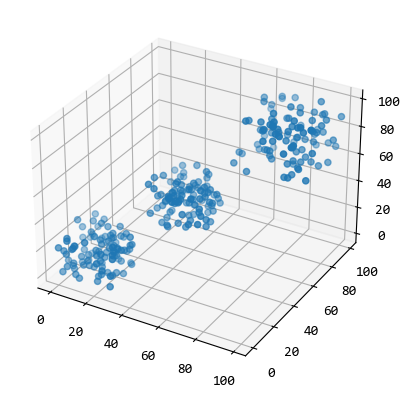
data1,data2,data3分佈在3個區域,
每個資料集有100條資料,每條資料有3個屬性。
1.1. 白化資料
聚類之前,一般會對資料進行白化,所謂白化資料,是指將資料集中的每個特徵或每個樣本的值都統一為同一個範圍。
這樣做的目的是為了消除特徵之間的量綱和數值大小差異,使得不同特徵具有相似的重要性,從而更容易進行聚類演演算法。
在聚類之前對資料進行白化處理也被稱為預處理階段。
from scipy.cluster.vq import whiten
# 白化資料
normal_data = whiten(data)
# 繪製白化後的資料
fig, ax = plt.subplots(subplot_kw={"projection": "3d"})
ax.scatter(normal_data[:, 0], normal_data[:, 1], normal_data[:, 2])
plt.show()
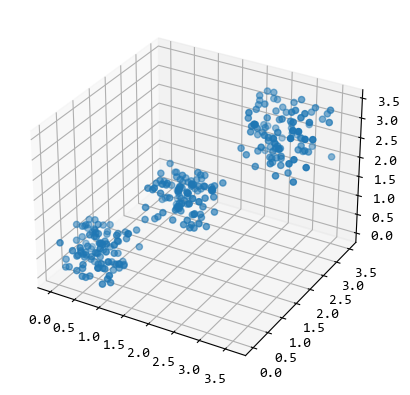
從圖中可以看出,資料的分佈情況沒有改變,只是資料的範圍從0~100變成0.0~3.5。
這就是白化的效果。
1.2. K-means
白化之後,就可以用K-meas方法來進行聚類運算了。scipy的vq模組中有2個聚類函數:kmeans和kmeans2。
kmeans函數最少只要傳入兩個引數即可:
- 需要聚類的資料,也就是上一步白化的資料
- 聚類的數目
返回值有2部分:
- 各個聚類的中心點
- 各個點距離聚類中心點的歐式距離的平均值
from scipy.cluster.vq import kmeans
center_points, distortion = kmeans(normal_data, 3)
print(center_points)
print(distortion)
# 執行結果
[[1.632802 1.56429847 1.51635413]
[0.48357948 0.55988559 0.48842058]
[2.81305235 2.84443275 2.78072325]]
0.5675874109728244
把三個聚類點繪製在圖中來看更加清楚:
fig, ax = plt.subplots(subplot_kw={"projection": "3d"})
ax.scatter(normal_data[:, 0],
normal_data[:, 1],
normal_data[:, 2])
ax.scatter(
center_points[:, 0],
center_points[:, 1],
center_points[:, 2],
color="r",
marker="^",
linewidths=5,
)
plt.show()
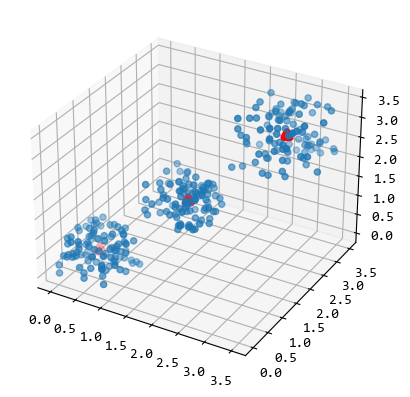
圖中3個紅色的點就是聚類的中心點。
1.3. K-means2
kmeans2函數使用起來和kmeans類似,但是返回值有區別,kmeans2的返回的是:
- 聚類的中心點座標
- 每個聚類中所有點的索引
from scipy.cluster.vq import kmeans2
center_points, labels = kmeans2(normal_data, 3)
print(center_points)
print(labels)
# 執行結果
[[2.81305235 2.84443275 2.78072325]
[1.632802 1.56429847 1.51635413]
[0.48357948 0.55988559 0.48842058]]
[2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
... ...
0 0 0 0]
可以看出,計算出的聚類中心點center_points和kmeans一樣(只是順序不一樣),labels有0,1,2三種值,代表normal_data中每個點屬於哪個分類。
kmeans2除了返回了聚類中心點,還有每個資料點屬於哪個聚類的資訊,
所以我們繪圖時,可以將屬於不同聚類的點標記不同的顏色。
fig, ax = plt.subplots(subplot_kw={"projection": "3d"})
arr_data = [[], [], []]
for idx, nd in enumerate(normal_data):
arr_data[labels[idx]].append(nd)
data = np.array(arr_data[0])
ax.scatter(data[:, 0], data[:, 1], data[:, 2], color='lightblue')
data = np.array(arr_data[1])
ax.scatter(data[:, 0], data[:, 1], data[:, 2], color='lightgreen')
data = np.array(arr_data[2])
ax.scatter(data[:, 0], data[:, 1], data[:, 2], color='lightyellow')
ax.scatter(
center_points[:, 0],
center_points[:, 1],
center_points[:, 2],
color="r",
marker="^",
linewidths=5,
)
plt.show()
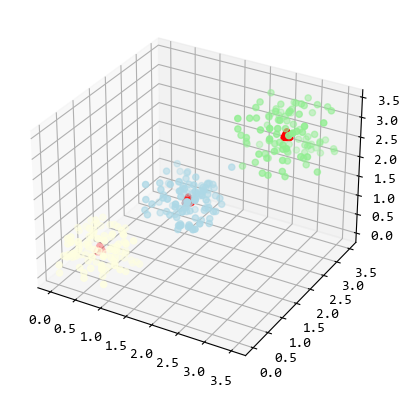
2. hierarchy 聚類
hierarchy聚類演演算法的步驟比較簡單:
- 將每個樣本視為一個簇
- 計算各個簇之間的距離,將距離最近的兩個簇合併為一個簇
- 重複第二個步驟,直至到最後一個簇
from scipy.cluster.hierarchy import ward, fcluster, dendrogram
from scipy.spatial.distance import pdist
# 計算樣本資料之間的距離
# normal_data是之前白化之後的資料
dist = pdist(normal_data)
# 在距離上建立Ward連線矩陣
Z = ward(dist)
# 層次聚類之後的平面聚類
S = fcluster(Z, t=0.9, criterion='distance')
print(S)
# 執行結果
[20 26 23 18 18 22 18 28 21 22 28 26 27 27 20 17 23 20 26 23 17 25 20 22
... ...
5 13 3 4 2 9 9 13 13 8 11 6]
返回的S中有300個資料,和normal_data中的資料一樣多,S中數值接近的點,分類越接近。
從數值看聚類結果不那麼明顯,scipy的層次聚類提供了一個dendrogram方法,內建了matpltlib的功能,
可以把層次聚類的結果用圖形展示出來。
P = dendrogram(Z, no_labels=True)
plt.show()
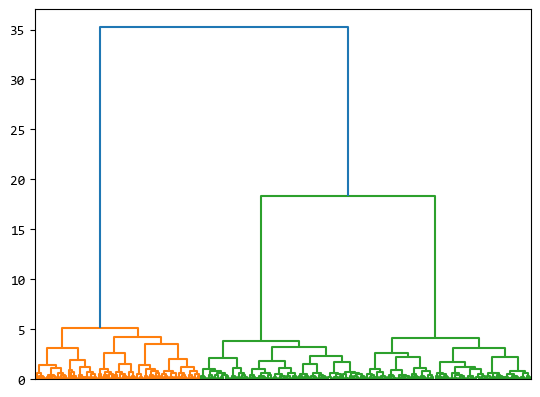
從這個圖可以看出每個資料分別屬於哪個層次的聚類。
最底層的葉子節點就是normal_datad中的各個資料,這些資料的索引資訊可以從 P 中獲取。
# P是一個字典,包含聚類之後的資訊
# key=ivl 是圖中最底層葉子節點在 normal_data 中的索引
print(P["ivl"])
# 執行結果
['236', '269', '244', ... ... '181', '175', '156', '157']
3. 總結
聚類分析可以幫助我們發現資料集中的內在結構、模式和相似性,從而更好地理解資料。
使用Scipy庫,可以幫助我們高效的完成資料的聚類分析,而不用去具體瞭解聚類分析演演算法的實現方式。