MindSpore簡要效能分析
技術背景
在之前的一篇部落格中,我們介紹過MindInsight的安裝與使用。不過在前面的文章中,我們主要介紹的是MindInsight與SummaryCollector的配合使用,更多的是用於對結果進行回溯。這篇文章我們簡要的從效能分析的角度,來介紹一下MindInsight的一些使用方法。
MindInsight的安裝與啟動
這部分內容在前面的部落格中已經介紹過一次,這裡簡單的重複一下相關的內容。安裝我們還是推薦使用pip進行安裝和管理:
$ python3 -m pip install mindinsight
啟動的方法很簡單,就是在指定的目錄下執行:
mindinsight start
如果在terminal裡面顯示如下內容,則表示安裝成功。
Web address: http://127.0.0.1:8080
service start state: success
使用Profiler分析運算元效能
當我們構建好相關的網路之後,類似於CPU中的line_profiler,這裡我們可以用MindSpore中所支援的Profiler來直接進行網路效能評估。這裡的Profiler主要以運算元為單位進行統計,最終會輸出每一個運算元的呼叫次數以及相關的佔用時長。使用方法非常簡單,就是在程式碼的開頭寫一句:profiler = ms.Profiler(start_profile=True),以及在結尾處寫一句:profiler.analyse()即可。
下面這個案例是MindSponge的一個能量極小化的案例。MindSponge是一個基於MindSpore框架開發的分子動力學模擬框架,更多的介紹和相關資訊可以參考MindSponge教學系列部落格。簡單來說,這裡我們只是模擬幾百個水分子的動力學演化過程。
import os
os.environ['GLOG_v']='4'
os.environ['MS_JIT_MODULES']='sponge'
import mindspore as ms
from mindspore import context
from mindspore.nn import Adam
if __name__ == "__main__":
import sys
sys.path.insert(0, '..')
from sponge import Sponge, Molecule, ForceField, WithEnergyCell
from sponge.callback import RunInfo
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
profiler = ms.Profiler(start_profile=True)
system = Molecule(template='water.tip3p.yaml')
system.set_pbc_box([0.4, 0.4, 0.4])
system.repeat_box([5, 5, 5])
potential = ForceField(system, parameters=['TIP3P'], use_pme=False)
opt = Adam(system.trainable_params(), 1e-3)
sim = WithEnergyCell(system, potential)
mini = Sponge(sim, optimizer=opt)
run_info = RunInfo(10)
mini.run(200, callbacks=[run_info])
profiler.analyse()
該模擬過程的輸出如下所示:
[MindSPONGE] Started simulation at 2023-09-11 10:57:03
[MindSPONGE] Compilation Time: 2.49s
[MindSPONGE] Step: 0, E_pot: 11003.434, Time: 2494.60ms
[MindSPONGE] Step: 10, E_pot: 9931.012, Time: 55.70ms
[MindSPONGE] Step: 20, E_pot: 9860.436, Time: 51.15ms
[MindSPONGE] Step: 30, E_pot: 9833.985, Time: 50.74ms
[MindSPONGE] Step: 40, E_pot: 9820.092, Time: 52.73ms
[MindSPONGE] Step: 50, E_pot: 9805.144, Time: 48.95ms
[MindSPONGE] Step: 60, E_pot: 9781.4375, Time: 47.87ms
[MindSPONGE] Step: 70, E_pot: 9740.486, Time: 48.46ms
[MindSPONGE] Step: 80, E_pot: 9684.311, Time: 48.67ms
[MindSPONGE] Step: 90, E_pot: 9619.269, Time: 52.02ms
[MindSPONGE] Step: 100, E_pot: 9555.06, Time: 51.39ms
[MindSPONGE] Step: 110, E_pot: 9498.154, Time: 47.72ms
[MindSPONGE] Step: 120, E_pot: 9450.13, Time: 49.52ms
[MindSPONGE] Step: 130, E_pot: 9408.7705, Time: 48.77ms
[MindSPONGE] Step: 140, E_pot: 9370.615, Time: 50.83ms
[MindSPONGE] Step: 150, E_pot: 9332.237, Time: 48.89ms
[MindSPONGE] Step: 160, E_pot: 9290.162, Time: 49.57ms
[MindSPONGE] Step: 170, E_pot: 9240.263, Time: 52.04ms
[MindSPONGE] Step: 180, E_pot: 9177.134, Time: 51.12ms
[MindSPONGE] Step: 190, E_pot: 9094.976, Time: 53.40ms
[MindSPONGE] Finished simulation at 2023-09-11 10:57:15
[MindSPONGE] Simulation time: 11.91 seconds.
--------------------------------------------------------------------------------
執行結束後,會在路徑下生成一個data/profiler/的目錄,裡面存放有我們所需的效能分析相關資訊。但是需要注意的是,profiler本身也會佔用很多的執行時間,所以使用profiler和不使用profiler的執行時間會有比較大的差別。我們如果只是希望對演演算法程式碼進行優化,只要保持在同一個條件(使用或不使用profiler)下進行分析即可。
MindInsight效能分析
如果在前面程式碼執行的路徑下直接啟動MindInsight,然後把http://127.0.0.1:8080複製到瀏覽器裡面開啟,就可以看到對應的效能資料,如下圖所示:
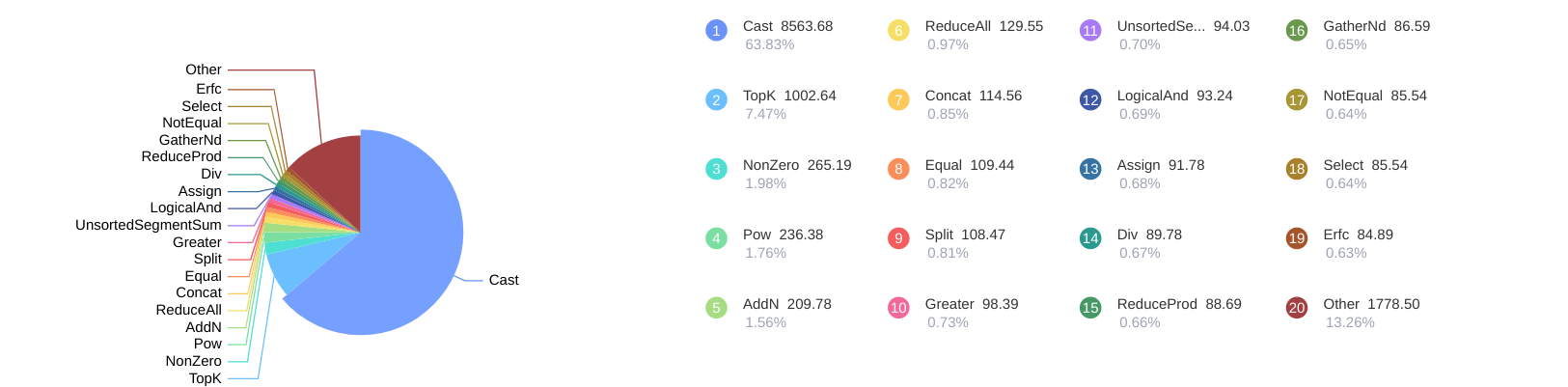
除了圖形化的資料展示之外,還可以在列表中逐項展開,去檢視每一個Operator的佔用時間:
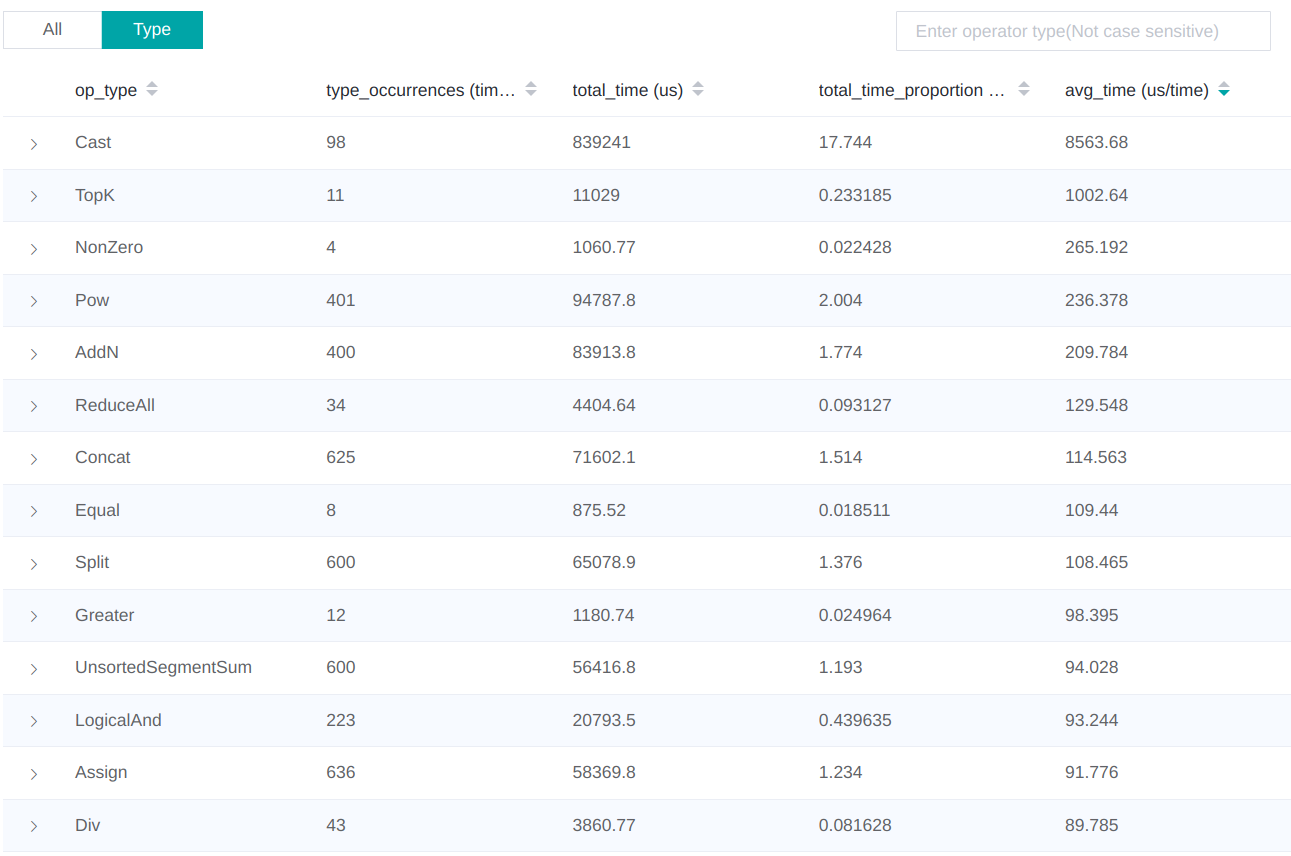
在這個結果中我們發現,由於使用了太多的Cast,有可能導致整體程式碼執行速度低下。如此一來,我們就可以挨個去查詢程式碼中所用到的Cast運算元,然後有針對性的進行優化:
$ grep -n -r "Cast" ~/projects/gitee/dechin/mindsponge/sponge/
/home/dechin/projects/gitee/dechin/mindsponge/sponge/potential/energy/coulomb.py:399: self.cast = ops.Cast()
/home/dechin/projects/gitee/dechin/mindsponge/sponge/potential/energy/coulomb.py:490: self.cast = ops.Cast()
$ grep -n -r "F.cast" ~/projects/gitee/dechin/mindsponge/sponge/
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/fullconnect.py:75: fc_idx = nrange + F.cast(no_idx <= nrange, ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/distance.py:142: max_neighbours = ops.count_nonzero(F.cast(mask, ms.float16), -1, dtype=ms.float16) - 1
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/distance.py:143: return F.cast(ops.reduce_max(max_neighbours), ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/distance.py:239: distances = F.cast(distances, ms.float16)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/distance.py:242: distances = F.cast(distances, ms.float32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/grids.py:324: sorted_grid_idx, sort_arg = self.sort(F.cast(atom_grid_idx, ms.float16))
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/grids.py:325: sorted_grid_idx = F.cast(sorted_grid_idx, ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/grids.py:356: grid_neigh_atoms, _ = self.sort(F.cast(grid_neigh_atoms, ms.float16))
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/grids.py:357: grid_neigh_atoms = F.cast(grid_neigh_atoms, ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/partition/grids.py:360: max_neighbours = F.cast(msnp.amax(F.cast(max_neighbours, ms.float32)), ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/metrics/metrics.py:455: classes_w_tensor = F.cast(classes_w_t2, mstype.float32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/metrics/metrics.py:482: classes_num = F.cast(classes_num, mstype.float32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/metrics/metrics.py:541: target = F.cast(target, mstype.float32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/metrics/metrics.py:542: probs = F.cast(prediction, mstype.float32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/function/operations.py:295: n = func.keepdims_sum(F.cast(mask, ms.int32), -2)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/function/functions.py:618: return F.cast(image, ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/function/functions.py:1409: value = F.cast(value, dtype)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/function/functions.py:1441: value = F.cast(value, dtype)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/system/residue/residue.py:262: self.natom_tensor = msnp.sum(F.cast(self.atom_mask, ms.float32), -1, keepdims=True)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/system/residue/residue.py:572: F.cast(self.atom_mask, ms.int32), -1, keepdims=True)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/system/molecule/molecule.py:633: self.system_natom = msnp.sum(F.cast(self.atom_mask, ms.float32), -1, keepdims=True)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/sampling/bias/metad.py:191: cutoff_bins = F.cast(cutoff_bins, ms.int32)
/home/dechin/projects/gitee/dechin/mindsponge/sponge/sampling/bias/metad.py:398: return F.cast(nearest_grid, ms.int32)
這裡優化的思路和過程我們暫時就不做展示了,本文主要介紹的是一個效能優化的思路:先用profiler定位到效能決速步,然後有針對性的進行優化。
MindInsight檢視計算圖
在使用AI框架進行計算的時候,我們的各種運算元會被編譯成一張大的計算圖,使用MindInsight就可以對這個計算圖進行視覺化。使用方法也很簡單,在設定context的時候多加上兩項設定即可:
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1,)
save_graphs=True, save_graphs_path='./graphs/')
需要留意的是,這個save_graphs_path一定要設定上,否則輸出的一大堆檔案在當前目錄下,直接沒眼看。接下來同樣的執行程式碼,會在當前目錄下生成一個graphs/資料夾。此時重新整理一下MindInsight的頁面,點選最上面的訓練列表,這個時候就會看到有兩個資料列:
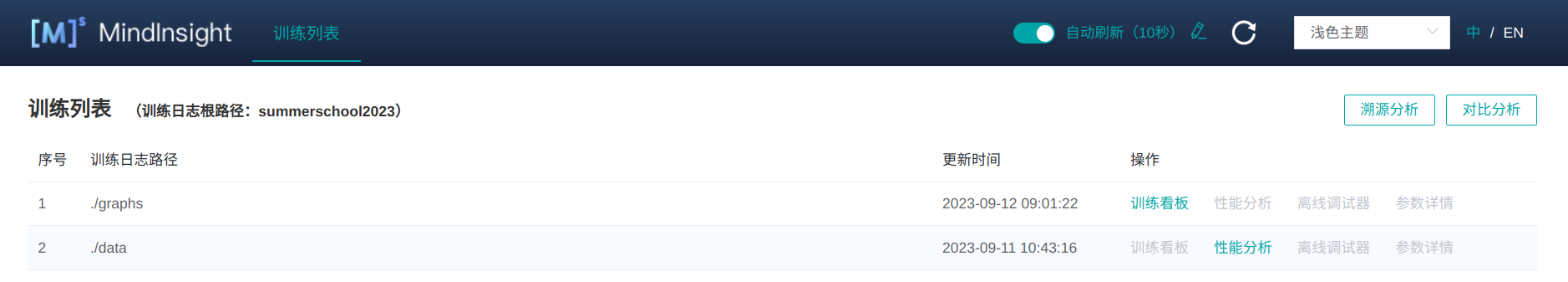
其中graphs這個資料列就是計算圖的內容,點進去以後的介面如下所示:
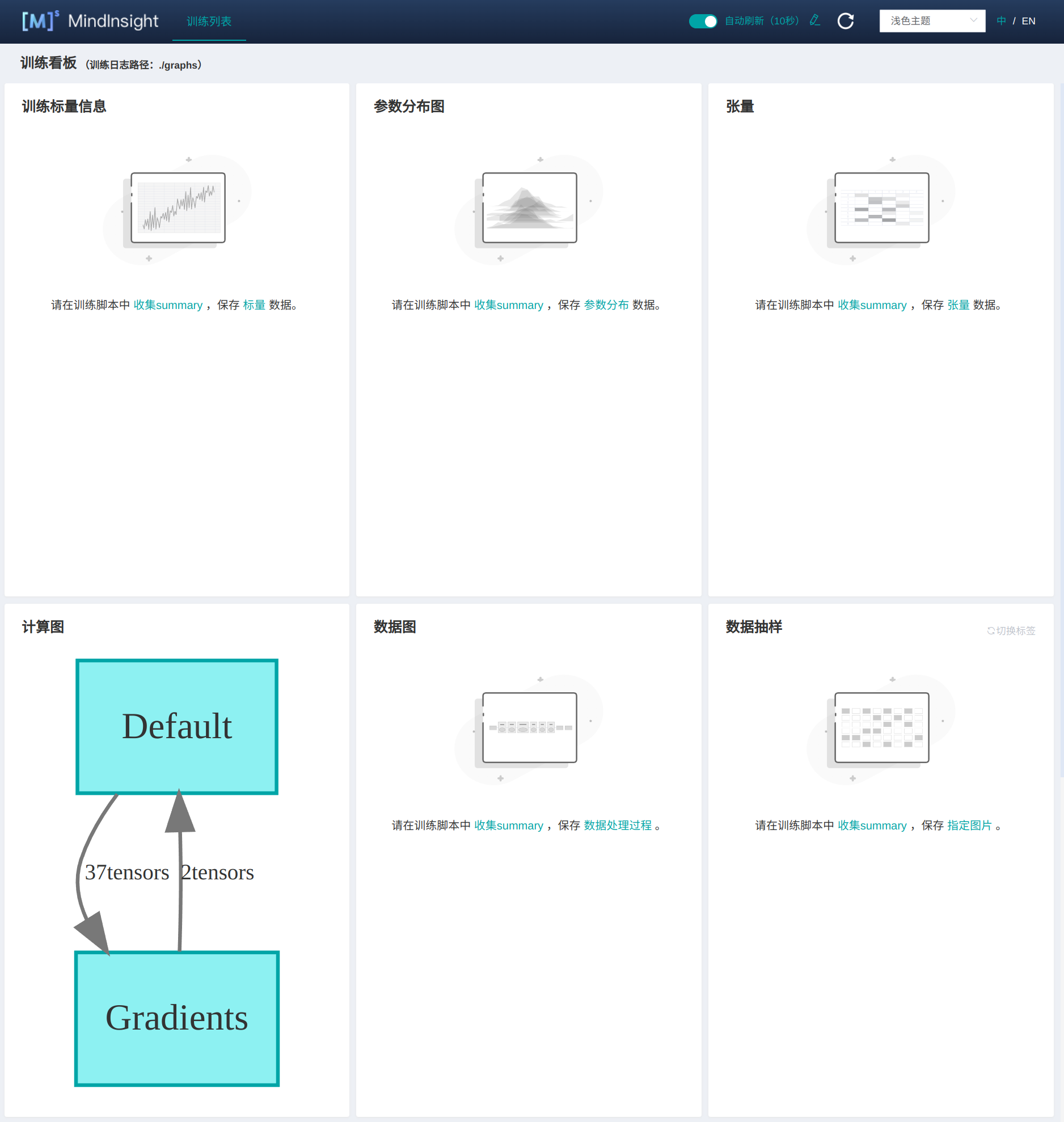
如果我們比較關注計算圖的話,就可以點進計算圖的介面:
這個計算圖的介面是根據右邊的目錄展開而展開的,如果我們想關注某一個模組的細節,就可以點進目錄樹:
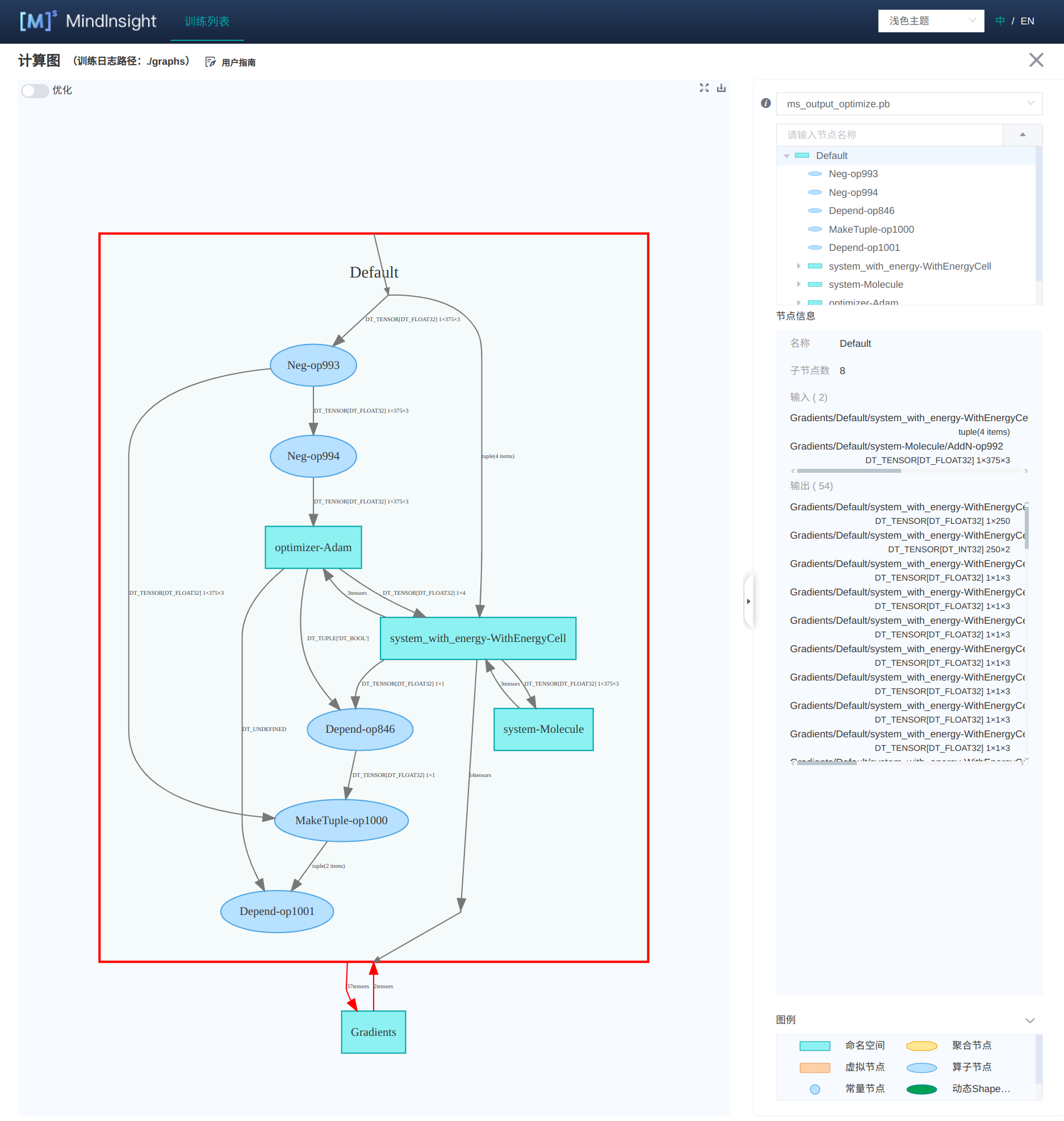
這樣的計算圖結構,便於大家對整體的效能進行調節和優化。
總結概要
當我們需要優化程式效能的時候,首先我們就需要了解程式的主要耗時模組在哪裡,也就是通常所謂的決速步,或者瓶頸模組,這樣就可以有針對性的去進行優化。在MindSpore相關的程式中,我們可以使用MindInsight這一強力的效能分析視覺化工具來進行分析。該工具會給出每個運算元的呼叫次數以及總耗時等引數,能夠給效能優化帶來不少重要的參考。
版權宣告
本文首發連結為:https://www.cnblogs.com/dechinphy/p/optimize.html
作者ID:DechinPhy
更多原著文章:https://www.cnblogs.com/dechinphy/
請博主喝咖啡:https://www.cnblogs.com/dechinphy/gallery/image/379634.html