MindSponge分子動力學模擬——Constraint約束(2023.09)
技術背景
在前面的幾篇部落格中,我們已經介紹了MindSponge的基本使用方法,比如定義一個分子系統、計算分子的單點能以及迭代器的使用等。有了這些基礎的教學,使用者以及可以執行一些比較簡單的模擬任務,比如可以跑一個能量極小化,或者是NVT過程。
當我們去執行一個模擬任務時,比較關鍵的一個指標是給定時間內可模擬的總時長,總時長直接決定了我們能不能看到其中的一些過渡態。當然,現在有很多改進的思路。比如可以使用硬體加速,縮減單步模擬的時長。比如可以使用增強取樣,朝著給定的方向演化,更容易看到中間態。本文要介紹的方法也是其中之一,可以採用Constraint約束的方法,限制分子體系中某些不重要的自由度,這樣演化步長\(dt\)就可以設定的大一些,也能夠起到加速的作用。常用的Constraint約束演演算法有Lincs和SETTLE演演算法。而Shake演演算法由於需要迭代,因此未在MindSponge中實現。
這裡還需要談一下Constraint和Restraint的區別。一般情況下,Constraint指代一些比較強硬的約束,比如SETTLE演演算法,直接就固定了一個三角形的形狀大小,在所有的演化過程中都不會發生變化。而Restraint更多的指代一些比較靈活的軟約束,比如加一個球形諧振子勢把分子系統限制在一個球體裡面,就是一種Restraint約束。而Restraint也並不全是軟約束,比如在MetaDynamics中會在邊界處施加一個梯度非常大的Restraint約束,雖然理論上是一個軟約束,但是CV一般無法越過這個邊界。
不施加約束
本案例類似於上一篇部落格中所介紹的NVT過程,不過上一篇部落格里面用的體系是一個純粹的多肽體系,這裡我們用Molecule自帶的加水的方法,模擬一個帶溶劑的多肽混合體系。使用的多肽是一個簡單的4個residue的體系:
REMARK Generated By Xponge (Molecule)
ATOM 1 N ALA 1 -0.095 -11.436 -0.780
ATOM 2 CA ALA 1 -0.171 -10.015 -0.507
ATOM 3 CB ALA 1 1.201 -9.359 -0.628
ATOM 4 C ALA 1 -1.107 -9.319 -1.485
ATOM 5 O ALA 1 -1.682 -9.960 -2.362
ATOM 6 N ARG 2 -1.303 -8.037 -1.397
ATOM 7 CA ARG 2 -2.194 -7.375 -2.328
ATOM 8 CB ARG 2 -3.606 -7.943 -2.235
ATOM 9 CG ARG 2 -4.510 -7.221 -3.228
ATOM 10 CD ARG 2 -5.923 -7.789 -3.136
ATOM 11 NE ARG 2 -6.831 -7.111 -4.087
ATOM 12 CZ ARG 2 -8.119 -7.421 -4.205
ATOM 13 NH1 ARG 2 -8.686 -8.371 -3.468
ATOM 14 NH2 ARG 2 -8.844 -6.747 -5.093
ATOM 15 C ARG 2 -2.273 -5.882 -2.042
ATOM 16 O ARG 2 -1.630 -5.388 -1.119
ATOM 17 N ALA 3 -3.027 -5.119 -2.777
ATOM 18 CA ALA 3 -3.103 -3.697 -2.505
ATOM 19 CB ALA 3 -1.731 -3.041 -2.625
ATOM 20 C ALA 3 -4.039 -3.001 -3.483
ATOM 21 O ALA 3 -4.614 -3.643 -4.359
ATOM 22 N ALA 4 -4.235 -1.719 -3.394
ATOM 23 CA ALA 4 -5.126 -1.057 -4.325
ATOM 24 CB ALA 4 -6.538 -1.625 -4.233
ATOM 25 C ALA 4 -5.205 0.436 -4.039
ATOM 26 O ALA 4 -4.561 0.930 -3.116
ATOM 27 OXT ALA 4 -5.915 1.166 -4.728
TER
具體的使用方法這裡就不多做介紹了,有相關的疑問的,可以在這個部落格列表中去尋找相關問題的答案。一個不包含約束的MindSponge分子動力學模擬程式碼如下:
from mindspore import nn, context
from sponge import ForceField, Sponge, set_global_units, Protein, UpdaterMD
from sponge.function import VelocityGenerator
from sponge.callback import RunInfo, WriteH5MD
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
set_global_units('A', 'kcal/mol')
# 載入一個純粹的多肽體系
system = Protein('../pdb/case1.pdb', template=['protein0.yaml'], rebuild_hydrogen=True)
# 在多肽體系的周邊填充厚度為4A的水分子,同時系統變為週期性重複的體系
system.fill_water(edge=4, template='water.tip3p.yaml')
# 載入兩個不同的力場,如果指定不使用PME演演算法,模擬速度會快一點,但存在能量偏移的問題,
# 而這裡只做展示用,所以暫時關閉PME演演算法的使用
energy = ForceField(system, parameters=['AMBER.FF99SB', 'TIP3P'], use_pme=False)
# 能量極小化過程,用的是MindSpore自帶的Adam優化器
min_opt = nn.Adam(system.trainable_params(), 1e-03)
md = Sponge(system, potential=energy, optimizer=min_opt)
run_info = RunInfo(200)
md.run(2000, callbacks=[run_info])
# 定義一個新的迭代器,並用change_optimizer完成切換
vgen = VelocityGenerator(300)
velocity = vgen(system.shape, system.atom_mass)
# 定義了NVT迭代器,指定300K的控溫
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin')
md.change_optimizer(opt)
nvt_h5md = WriteH5MD(system, 'test_nvt.h5md', save_freq=10, write_image=False)
md.run(2000, callbacks=[run_info, nvt_h5md])
該程式碼的執行結果如下:
[MindSPONGE] Adding 45 hydrogen atoms for the protein molecule in 0.003 seconds.
[MindSPONGE] The box size used when filling water is: [21.184929 22.856928 15.495929]
[MindSPONGE] The edge gap along x axis is 4.0.
[MindSPONGE] The edge gap along y axis is 3.999999523162842.
[MindSPONGE] The edge gap along z axis is 3.999999761581421.
[MindSPONGE] Totally 172 waters is added to the system!
[MindSPONGE] Adding water molecule task finished!
[MindSPONGE] Started simulation at 2023-09-05 15:31:31
[MindSPONGE] Step: 0, E_pot: 6019.2305
[MindSPONGE] Step: 200, E_pot: 3667.4219
[MindSPONGE] Step: 400, E_pot: 3007.9343
[MindSPONGE] Step: 600, E_pot: 2633.8374
[MindSPONGE] Step: 800, E_pot: 2380.708
[MindSPONGE] Step: 1000, E_pot: 2233.6882
[MindSPONGE] Step: 1200, E_pot: 2129.8638
[MindSPONGE] Step: 1400, E_pot: 2050.8691
[MindSPONGE] Step: 1600, E_pot: 1993.4315
[MindSPONGE] Step: 1800, E_pot: 1949.2118
[MindSPONGE] Finished simulation at 2023-09-05 15:32:19
[MindSPONGE] Simulation time: 47.67 seconds.
--------------------------------------------------------------------------------
[MindSPONGE] Started simulation at 2023-09-05 15:32:20
[MindSPONGE] Step: 0, E_pot: 1910.4984, E_kin: 540.1826, E_tot: 2450.6812, Temperature: 316.81873, Pressure: -2254.547, Volume: 7503.4756
[MindSPONGE] Step: 200, E_pot: 2165.2405, E_kin: 421.29523, E_tot: 2586.5356, Temperature: 247.09091, Pressure: -4248.9507, Volume: 7503.4756
[MindSPONGE] Step: 400, E_pot: 2167.1772, E_kin: 479.70053, E_tot: 2646.8777, Temperature: 281.34576, Pressure: -5078.793, Volume: 7503.4756
[MindSPONGE] Step: 600, E_pot: 2196.748, E_kin: 480.39355, E_tot: 2677.1416, Temperature: 281.75226, Pressure: -2657.5989, Volume: 7503.4756
[MindSPONGE] Step: 800, E_pot: 2191.3103, E_kin: 498.1831, E_tot: 2689.4934, Temperature: 292.18588, Pressure: 506.58923, Volume: 7503.4756
[MindSPONGE] Step: 1000, E_pot: 2203.0522, E_kin: 490.20367, E_tot: 2693.2559, Temperature: 287.5059, Pressure: -3832.6213, Volume: 7503.4756
[MindSPONGE] Step: 1200, E_pot: 2205.9346, E_kin: 475.74023, E_tot: 2681.6748, Temperature: 279.02304, Pressure: -3744.449, Volume: 7503.4756
[MindSPONGE] Step: 1400, E_pot: 2191.0286, E_kin: 502.4231, E_tot: 2693.4517, Temperature: 294.67264, Pressure: -3162.2998, Volume: 7503.4756
[MindSPONGE] Step: 1600, E_pot: 2200.712, E_kin: 511.2496, E_tot: 2711.9614, Temperature: 299.84943, Pressure: -2348.196, Volume: 7503.4756
[MindSPONGE] Step: 1800, E_pot: 2209.8, E_kin: 514.364, E_tot: 2724.164, Temperature: 301.67603, Pressure: -2295.4656, Volume: 7503.4756
[MindSPONGE] Finished simulation at 2023-09-05 15:33:25
[MindSPONGE] Simulation time: 1 minutes 5.4 seconds.
--------------------------------------------------------------------------------
需要注意的是,雖然我們在補水的時候載入的模板(如下所示)裡面有SETTLE約束演演算法相關的設定資訊,但是因為在UpdaterMD中未指定constraint引數,因此預設也是不新增約束條件的。
template:
base: water_3p.yaml
WAT:
atom_charge: [-0.834, 0.417, 0.417]
settle:
mandatory: false
length_unit: nm
distance:
OW-HW: 0.09572
HW-HW: 0.15139
molecule:
residue:
- WAT
length_unit: nm
coordinate:
- [0.0, 0.0, 0.0]
- [0.079079641, 0.061207927, 0.0]
- [-0.079079641, 0.061207927, 0.0]
可以通過VMD外掛來檢視執行的軌跡,我們可以從中發現,因為未新增任何的約束條件,因此軌跡中的各個鍵長鍵角都在不斷的變化。
水分子SETTLE約束
在上一個章節中,我們所執行的體系是一個多肽和水溶液的混合體系,並且沒有施加任何的約束條件。這裡我們先介紹第一種約束條件,對水分子施加一個SETTLE約束,簡單來說,就是固定住水分子的三個原子所形成的三角形。其實這個約束更多的是在TIP4P力場中發揮作用,不過在2023年9月份的當前MindSponge版本,暫不支援TIP4P力場。
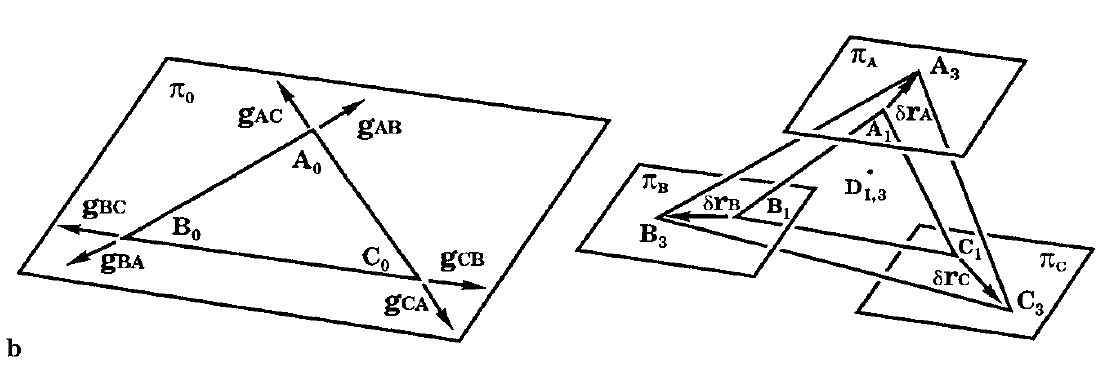
這裡先簡要介紹一下SETTLE約束演演算法的原理(如上圖所示),方便大家瞭解SETTLE約束演演算法的適用範圍。三角形\(A_0B_0C_0\)是原始的三角形,三角形\(A_1B_1C_1\)是在力場作用下移動了一步的三角形。我們的目標,是對新的三角形的三個頂點進行平移變換,得到一個符合約束條件(SETTLE演演算法的約束條件就是三個邊長引數,OW-HW和HW-HW要等於給定值)的結果。而新三角形的三個點\(A_1,B_1,C_1\),只能夠在原始三角形平面的切向移動,移動後找到一個符合邊長約束條件的三角形\(A_3B_3C_3\)作為最終的結果。
由此可見,SETTLE約束演演算法的適用範圍,只能作用於三角形的體系,且必須是等腰三角形。在MindSponge中適用SETTLE約束演演算法的方法非常簡單,就是在Updater迭代器的引數中新增一個constraint的引數即可,把構造好的SETTLE物件傳進去。當然,也可以直接從模板檔案裡面載入SETTLE物件,這個後面章節會提到。呼叫SETTLE演演算法相關的程式碼實現如下:
from mindspore import nn, context
from sponge import ForceField, Sponge, set_global_units, Protein, UpdaterMD
from sponge.function import VelocityGenerator
from sponge.control import SETTLE
from sponge.callback import RunInfo, WriteH5MD
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
set_global_units('A', 'kcal/mol')
system = Protein('../pdb/case1.pdb', template=['protein0.yaml'], rebuild_hydrogen=True)
system.fill_water(edge=4, template='water.tip3p.yaml')
energy = ForceField(system, parameters=['AMBER.FF99SB', 'TIP3P'], use_pme=False)
min_opt = nn.Adam(system.trainable_params(), 1e-03)
md = Sponge(system, potential=energy, optimizer=min_opt)
run_info = RunInfo(200)
md.run(2000, callbacks=[run_info])
vgen = VelocityGenerator(300)
velocity = vgen(system.shape, system.atom_mass)
# 在迭代器中傳入SETTLE物件
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin',
constraint=SETTLE(system))
md.change_optimizer(opt)
nvt_h5md = WriteH5MD(system, 'test_nvt.h5md', save_freq=10, write_image=False)
md.run(2000, callbacks=[run_info, nvt_h5md])
執行結果如下所示:
[MindSPONGE] Adding 45 hydrogen atoms for the protein molecule in 0.003 seconds.
[MindSPONGE] The box size used when filling water is: [21.184929 22.856928 15.495929]
[MindSPONGE] The edge gap along x axis is 4.0.
[MindSPONGE] The edge gap along y axis is 3.999999523162842.
[MindSPONGE] The edge gap along z axis is 3.999999761581421.
[MindSPONGE] Totally 172 waters is added to the system!
[MindSPONGE] Adding water molecule task finished!
[MindSPONGE] Started simulation at 2023-09-05 16:05:08
[MindSPONGE] Step: 0, E_pot: 6019.2305
[MindSPONGE] Step: 200, E_pot: 3667.421
[MindSPONGE] Step: 400, E_pot: 3007.9253
[MindSPONGE] Step: 600, E_pot: 2633.8325
[MindSPONGE] Step: 800, E_pot: 2380.7039
[MindSPONGE] Step: 1000, E_pot: 2233.6877
[MindSPONGE] Step: 1200, E_pot: 2129.8643
[MindSPONGE] Step: 1400, E_pot: 2050.8638
[MindSPONGE] Step: 1600, E_pot: 1993.42
[MindSPONGE] Step: 1800, E_pot: 1949.1956
[MindSPONGE] Finished simulation at 2023-09-05 16:05:53
[MindSPONGE] Simulation time: 45.13 seconds.
--------------------------------------------------------------------------------
[MindSPONGE] The settle constraint is used for the molecule system.
[MindSPONGE] Started simulation at 2023-09-05 16:05:54
[MindSPONGE] Step: 0, E_pot: 1910.4838, E_kin: 517.4474, E_tot: 2427.9312, Temperature: 303.48444, Pressure: -813660.7, Volume: 7503.4756
[MindSPONGE] Step: 200, E_pot: 2011.8678, E_kin: 235.43576, E_tot: 2247.3035, Temperature: 197.4598, Pressure: -13826140.0, Volume: 7503.4756
[MindSPONGE] Step: 400, E_pot: 1969.1597, E_kin: 229.6954, E_tot: 2198.855, Temperature: 192.64537, Pressure: -13705921.0, Volume: 7503.4756
[MindSPONGE] Step: 600, E_pot: 1941.4747, E_kin: 224.78912, E_tot: 2166.264, Temperature: 188.53047, Pressure: -13909719.0, Volume: 7503.4756
[MindSPONGE] Step: 800, E_pot: 1930.0374, E_kin: 207.89899, E_tot: 2137.9363, Temperature: 174.36473, Pressure: -14122749.0, Volume: 7503.4756
[MindSPONGE] Step: 1000, E_pot: 1916.6355, E_kin: 199.95618, E_tot: 2116.5918, Temperature: 167.7031, Pressure: -14373733.0, Volume: 7503.4756
[MindSPONGE] Step: 1200, E_pot: 1902.2065, E_kin: 203.48846, E_tot: 2105.695, Temperature: 170.66563, Pressure: -14358520.0, Volume: 7503.4756
[MindSPONGE] Step: 1400, E_pot: 1895.2224, E_kin: 218.30115, E_tot: 2113.5234, Temperature: 183.089, Pressure: -13955541.0, Volume: 7503.4756
[MindSPONGE] Step: 1600, E_pot: 1878.3259, E_kin: 198.46277, E_tot: 2076.7886, Temperature: 166.45056, Pressure: -14178873.0, Volume: 7503.4756
[MindSPONGE] Step: 1800, E_pot: 1880.648, E_kin: 200.18999, E_tot: 2080.838, Temperature: 167.8992, Pressure: -14218600.0, Volume: 7503.4756
[MindSPONGE] Finished simulation at 2023-09-05 16:08:00
[MindSPONGE] Simulation time: 2 minutes 6.1 seconds.
--------------------------------------------------------------------------------
到這裡我們還看不出什麼比較特別的結論,但是我們可以從VMD視覺化的結果中看看,相比於什麼約束都不加,有什麼樣的效果。
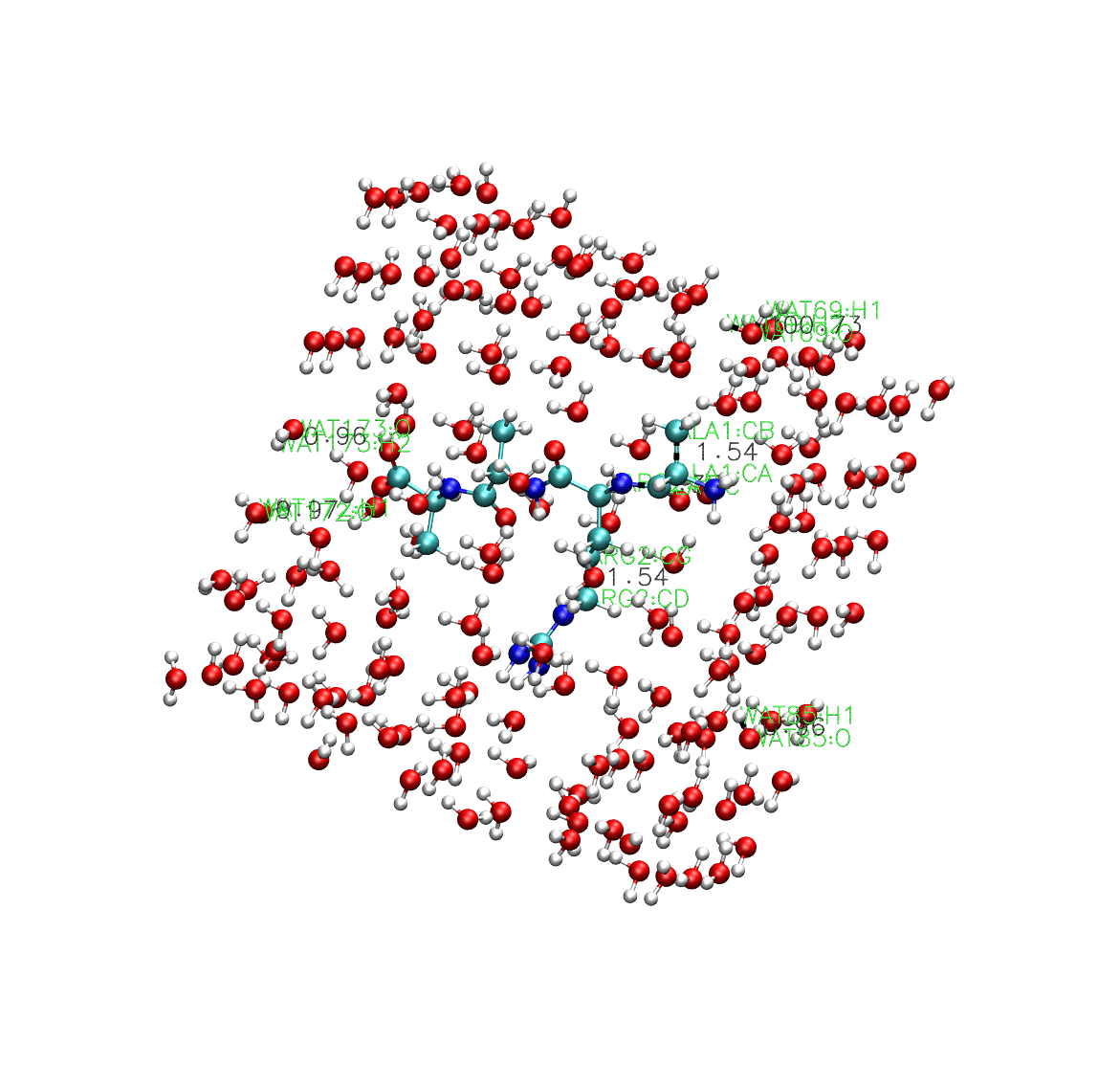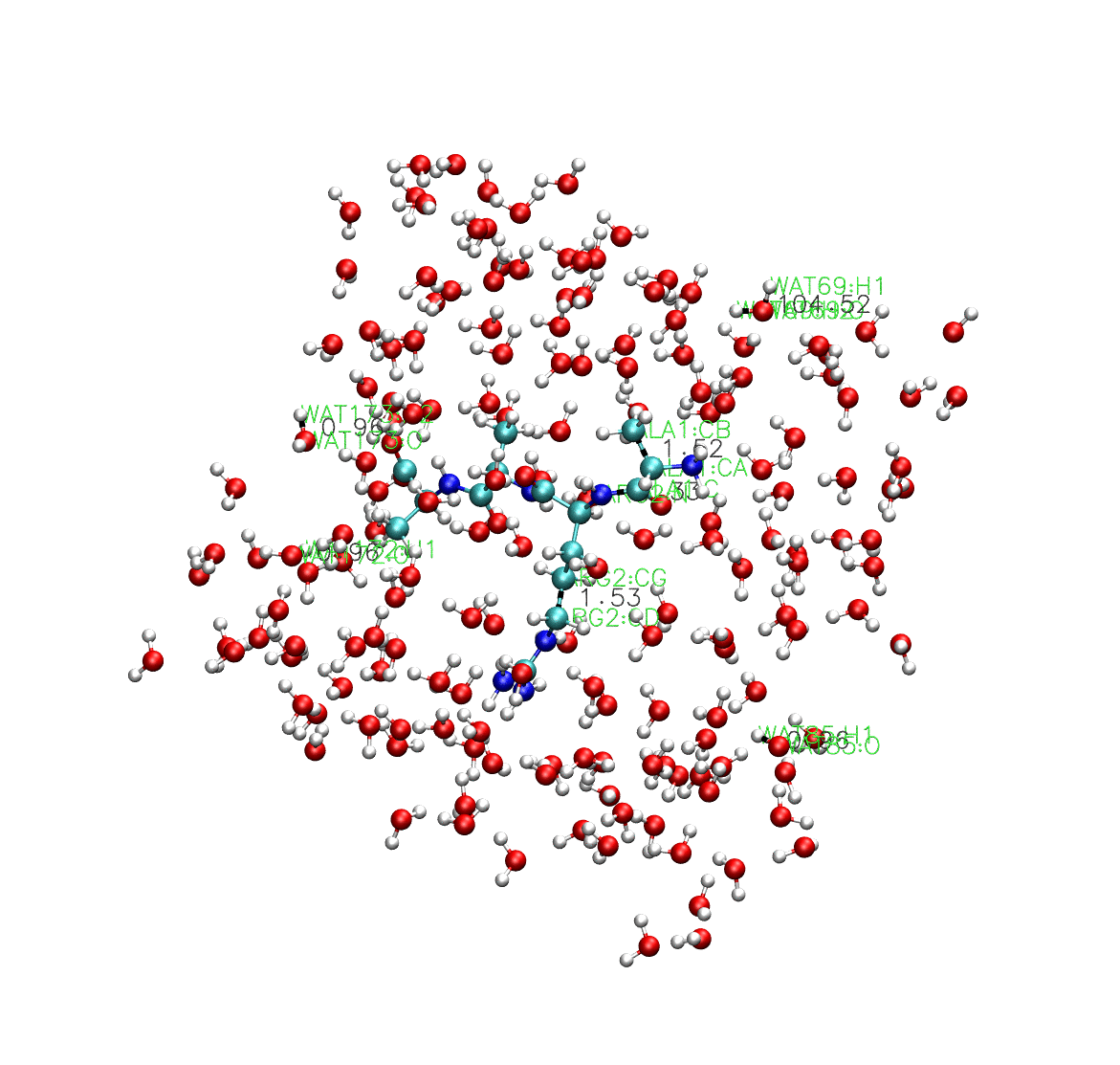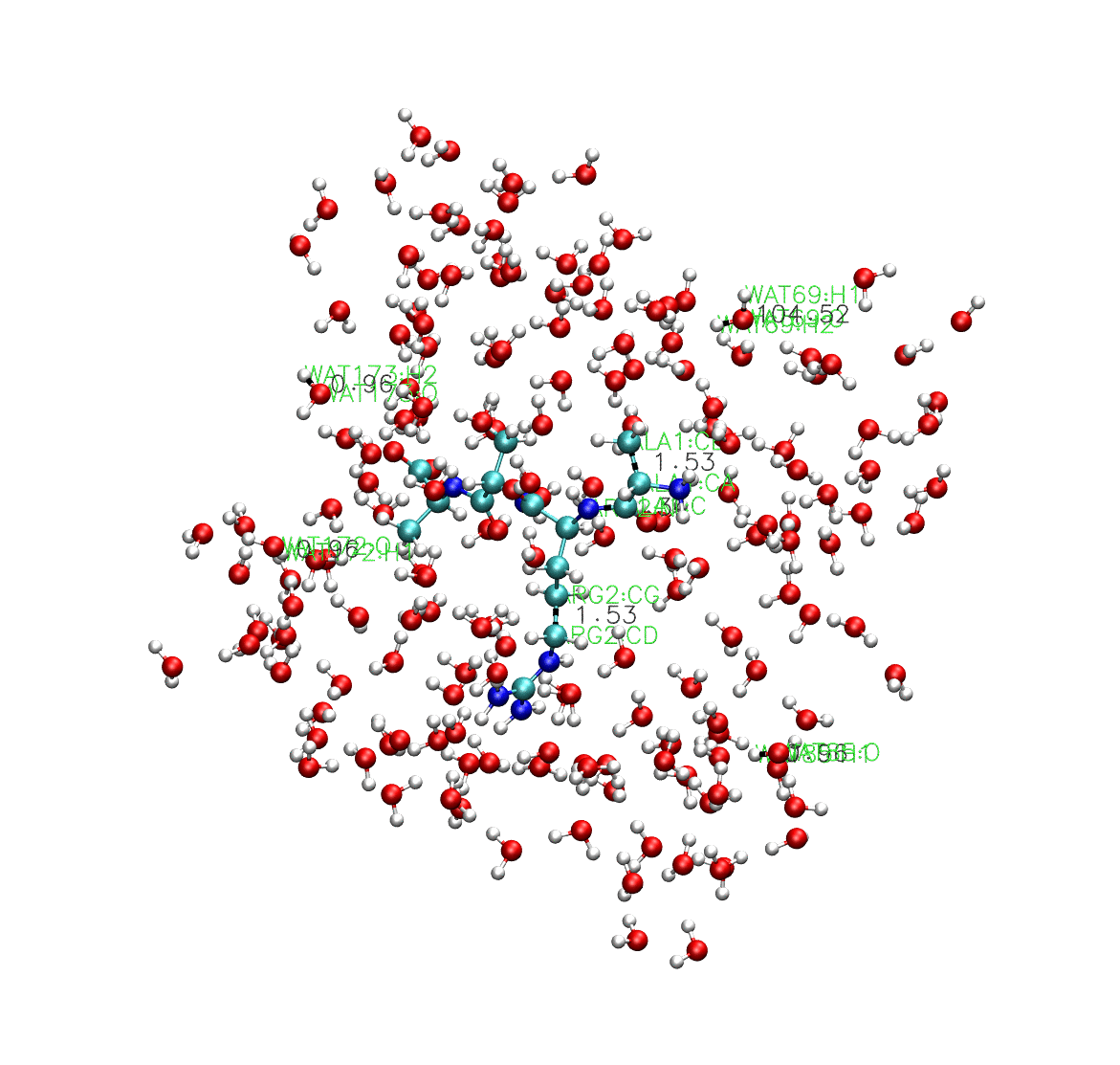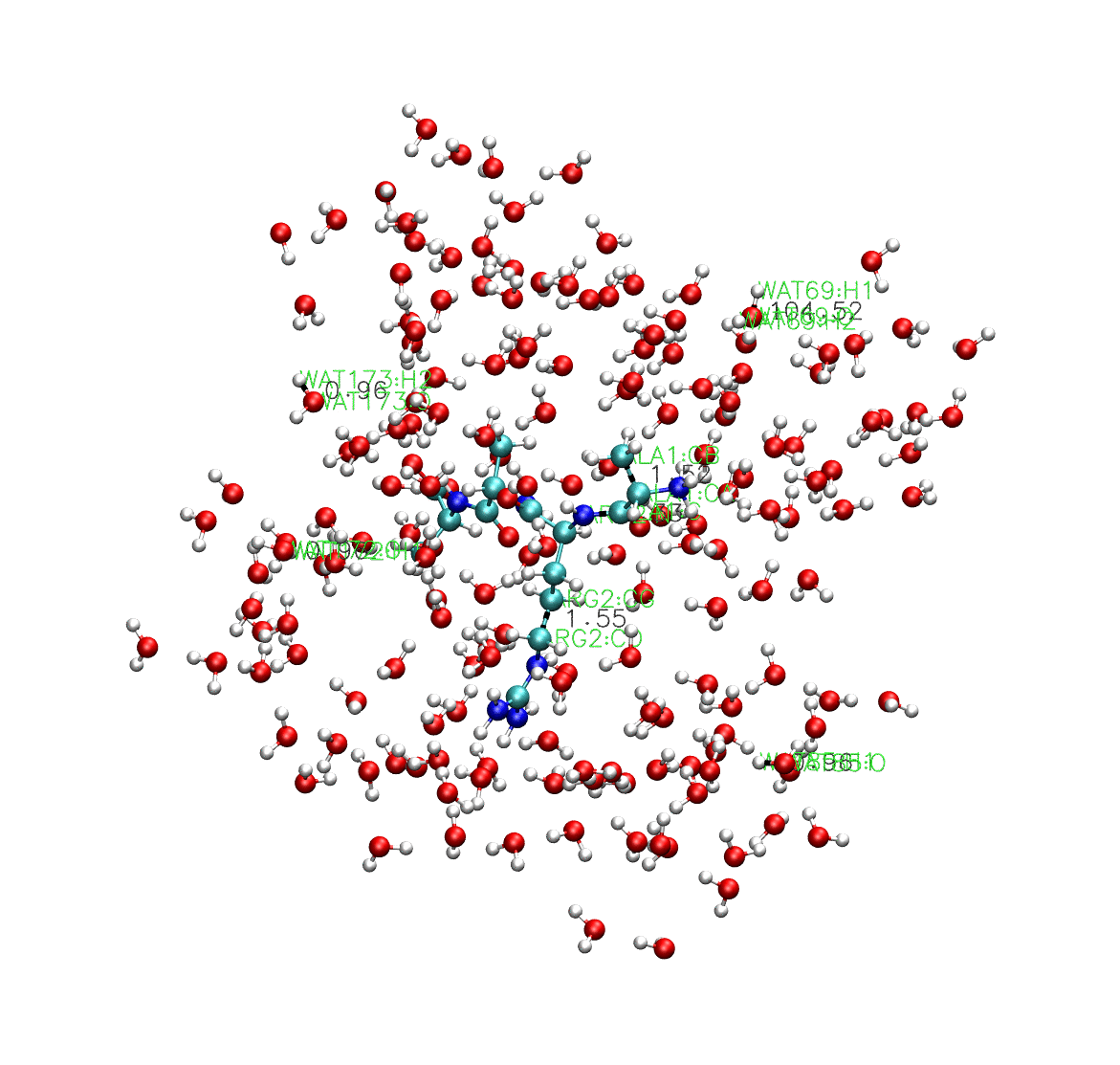
可以看到,在上面這個動態圖中,所有的水分子都在SETTLE約束演演算法的作用下保持鍵長鍵角不動,而中間的多肽因為不受SETTLE約束演演算法的影響,鍵長鍵角都在發生變化。
多肽Lincs約束
Lincs是一個只用於約束指定鍵的鍵長的約束演演算法,原理上相對複雜一點,可以參考下這篇部落格中的介紹。
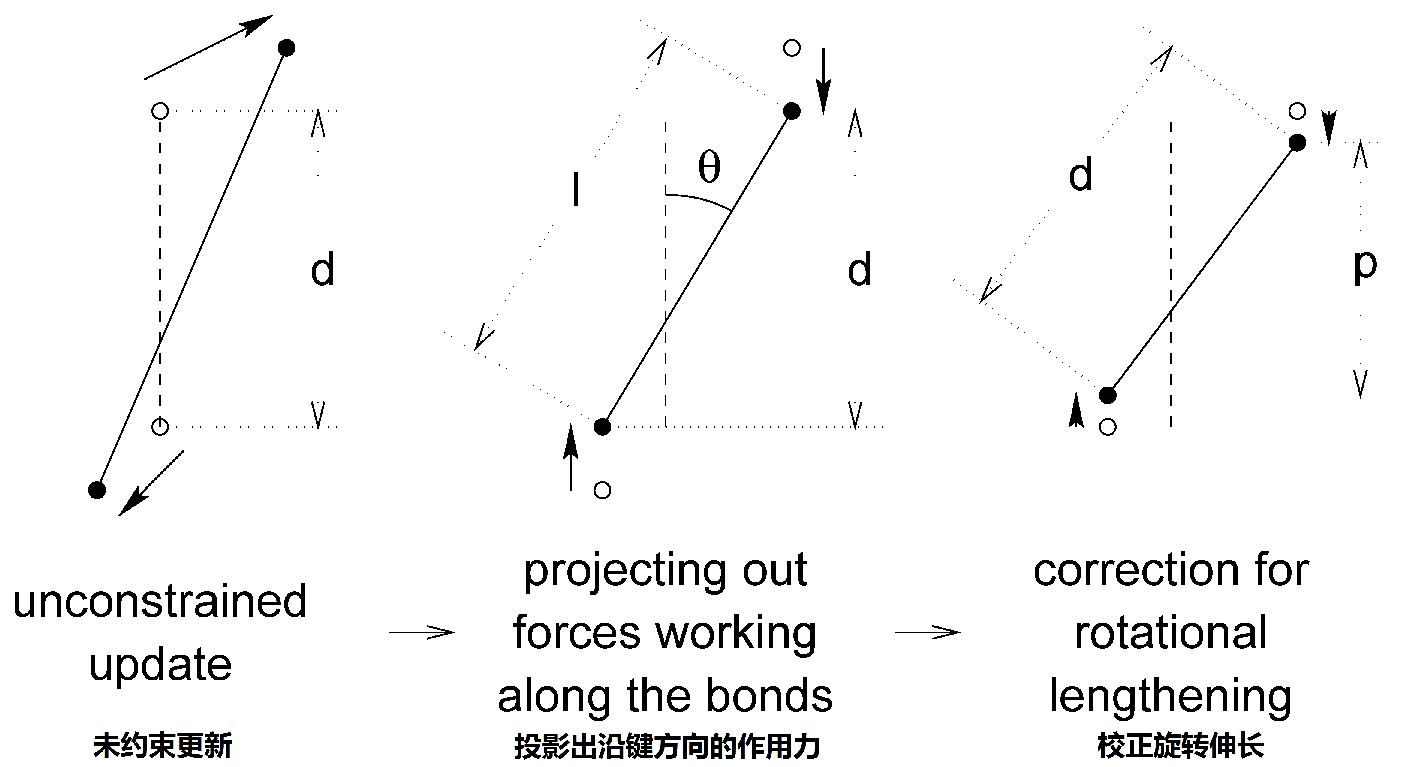
整體思路就像上面這張圖一樣,一根鍵在迭代器的作用下可能會發生偏移,此時在演化後的方向上把鍵長拉到指定的長度,這樣就完成了約束演演算法。我們也可以理解為,每一個鍵都伸縮一下,最後再拼起來變成一個新的狀態。這種約束演演算法,更多的適用於蛋白質的體系。一方面達不到TIP4P模型所需要的結果,另一方面Lincs演演算法本身是無法並行化和模組化的,效能也比不上SETTLE約束演演算法。因此對於水溶液體系裡面一般是不用Lincs約束的。
設定Lincs約束演演算法的方法跟SETTLE演演算法是一致的,只需要在UpdaterMD中傳入一個Lincs物件的constraint引數即可:
from mindspore import nn, context
from sponge import ForceField, Sponge, set_global_units, Protein, UpdaterMD
from sponge.function import VelocityGenerator
from sponge.control import Lincs
from sponge.callback import RunInfo, WriteH5MD
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
set_global_units('A', 'kcal/mol')
system = Protein('../pdb/case1.pdb', template=['protein0.yaml'], rebuild_hydrogen=True)
system.fill_water(edge=4, template='water.tip3p.yaml')
energy = ForceField(system, parameters=['AMBER.FF99SB', 'TIP3P'], use_pme=False)
min_opt = nn.Adam(system.trainable_params(), 1e-03)
md = Sponge(system, potential=energy, optimizer=min_opt)
run_info = RunInfo(200)
md.run(2000, callbacks=[run_info])
vgen = VelocityGenerator(300)
velocity = vgen(system.shape, system.atom_mass)
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin',
constraint=Lincs(system, 'h-bonds'))
md.change_optimizer(opt)
nvt_h5md = WriteH5MD(system, 'test_nvt.h5md', save_freq=10, write_image=False)
md.run(2000, callbacks=[run_info, nvt_h5md])
這裡的Lincs約束僅僅施加在氫鍵上。由於這是一個多肽和水分子的混合體系,實際上這裡的Lincs只施加在多肽上面的氫鍵(共價鍵)。上述程式碼的執行結果如下:
[MindSPONGE] Adding 45 hydrogen atoms for the protein molecule in 0.003 seconds.
[MindSPONGE] The box size used when filling water is: [21.184929 22.856928 15.495929]
[MindSPONGE] The edge gap along x axis is 4.0.
[MindSPONGE] The edge gap along y axis is 3.999999523162842.
[MindSPONGE] The edge gap along z axis is 3.999999761581421.
[MindSPONGE] Totally 172 waters is added to the system!
[MindSPONGE] Adding water molecule task finished!
[MindSPONGE] Started simulation at 2023-09-05 17:14:21
[MindSPONGE] Step: 0, E_pot: 6019.2305
[MindSPONGE] Step: 200, E_pot: 3667.4224
[MindSPONGE] Step: 400, E_pot: 3007.935
[MindSPONGE] Step: 600, E_pot: 2633.8376
[MindSPONGE] Step: 800, E_pot: 2380.7083
[MindSPONGE] Step: 1000, E_pot: 2233.6897
[MindSPONGE] Step: 1200, E_pot: 2129.8662
[MindSPONGE] Step: 1400, E_pot: 2050.8708
[MindSPONGE] Step: 1600, E_pot: 1993.4323
[MindSPONGE] Step: 1800, E_pot: 1949.2124
[MindSPONGE] Finished simulation at 2023-09-05 17:15:06
[MindSPONGE] Simulation time: 45.48 seconds.
--------------------------------------------------------------------------------
[MindSPONGE] The lincs constraint is used for the molecule system.
[MindSPONGE] Started simulation at 2023-09-05 17:15:07
[MindSPONGE] Step: 0, E_pot: 1910.498, E_kin: 515.0567, E_tot: 2425.5547, Temperature: 302.08228, Pressure: -1184091.6, Volume: 7503.4756
[MindSPONGE] Step: 200, E_pot: 2150.7756, E_kin: 402.22913, E_tot: 2553.005, Temperature: 240.10625, Pressure: -33554.688, Volume: 7503.4756
[MindSPONGE] Step: 400, E_pot: 2175.2153, E_kin: 444.4759, E_tot: 2619.6912, Temperature: 265.32498, Pressure: -80184.71, Volume: 7503.4756
[MindSPONGE] Step: 600, E_pot: 2186.1191, E_kin: 471.45215, E_tot: 2657.5713, Temperature: 281.42816, Pressure: -27345.148, Volume: 7503.4756
[MindSPONGE] Step: 800, E_pot: 2189.4526, E_kin: 504.2351, E_tot: 2693.6877, Temperature: 300.99756, Pressure: -75278.32, Volume: 7503.4756
[MindSPONGE] Step: 1000, E_pot: 2196.2683, E_kin: 522.76526, E_tot: 2719.0337, Temperature: 312.05896, Pressure: -38718.61, Volume: 7503.4756
[MindSPONGE] Step: 1200, E_pot: 2217.1887, E_kin: 489.45416, E_tot: 2706.6428, Temperature: 292.17426, Pressure: -59596.75, Volume: 7503.4756
[MindSPONGE] Step: 1400, E_pot: 2178.505, E_kin: 527.1349, E_tot: 2705.6396, Temperature: 314.66736, Pressure: -36722.918, Volume: 7503.4756
[MindSPONGE] Step: 1600, E_pot: 2161.2007, E_kin: 501.55945, E_tot: 2662.7603, Temperature: 299.4004, Pressure: -33997.46, Volume: 7503.4756
[MindSPONGE] Step: 1800, E_pot: 2185.1304, E_kin: 493.74838, E_tot: 2678.8787, Temperature: 294.73767, Pressure: -55893.01, Volume: 7503.4756
[MindSPONGE] Finished simulation at 2023-09-05 17:16:24
[MindSPONGE] Simulation time: 1 minutes 16.8 seconds.
--------------------------------------------------------------------------------
從列印的輸出結果中我們可以發現,這裡lincs constraint被呼叫到了。同樣的,我們可以用VMD外掛對結果進行視覺化:
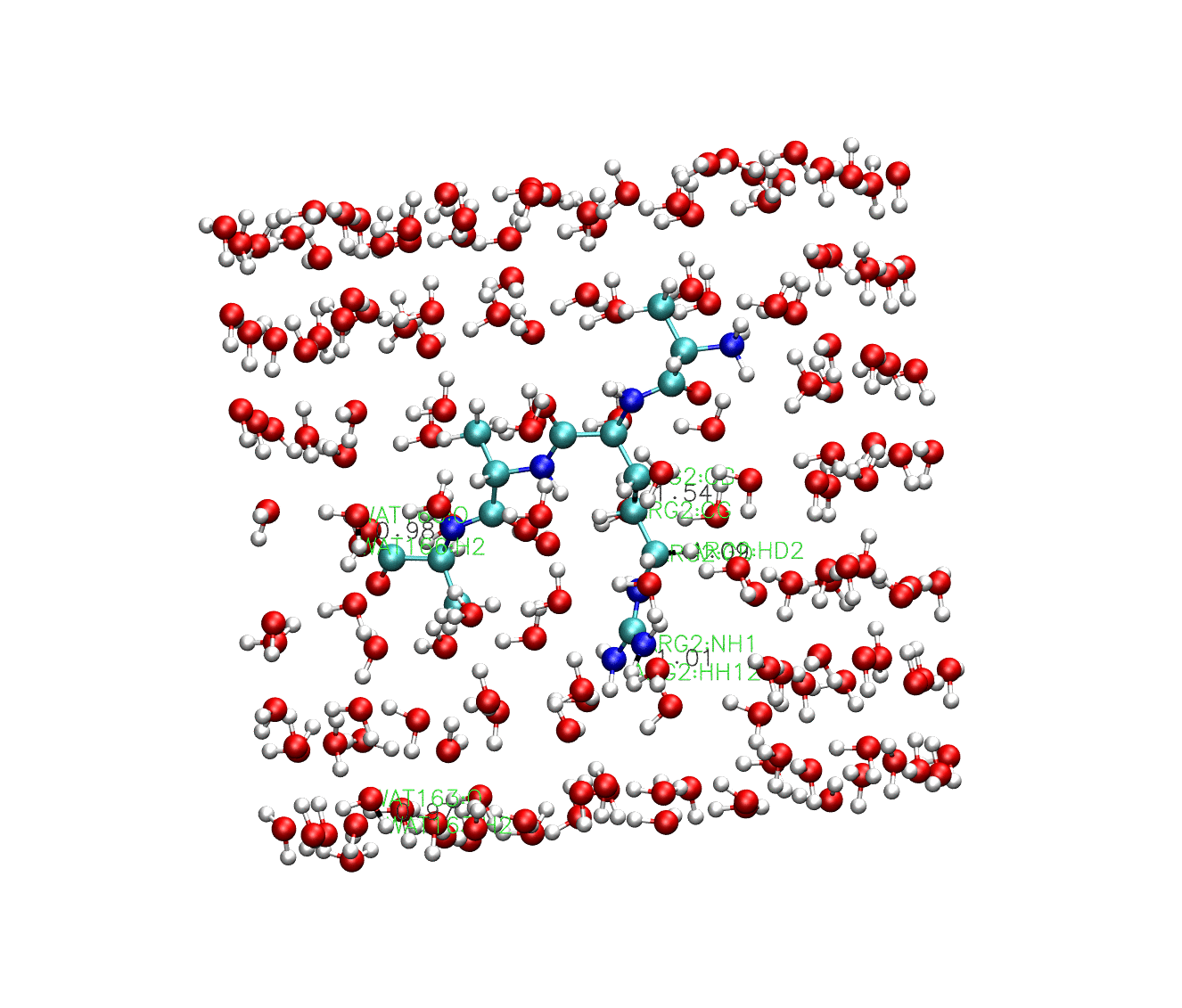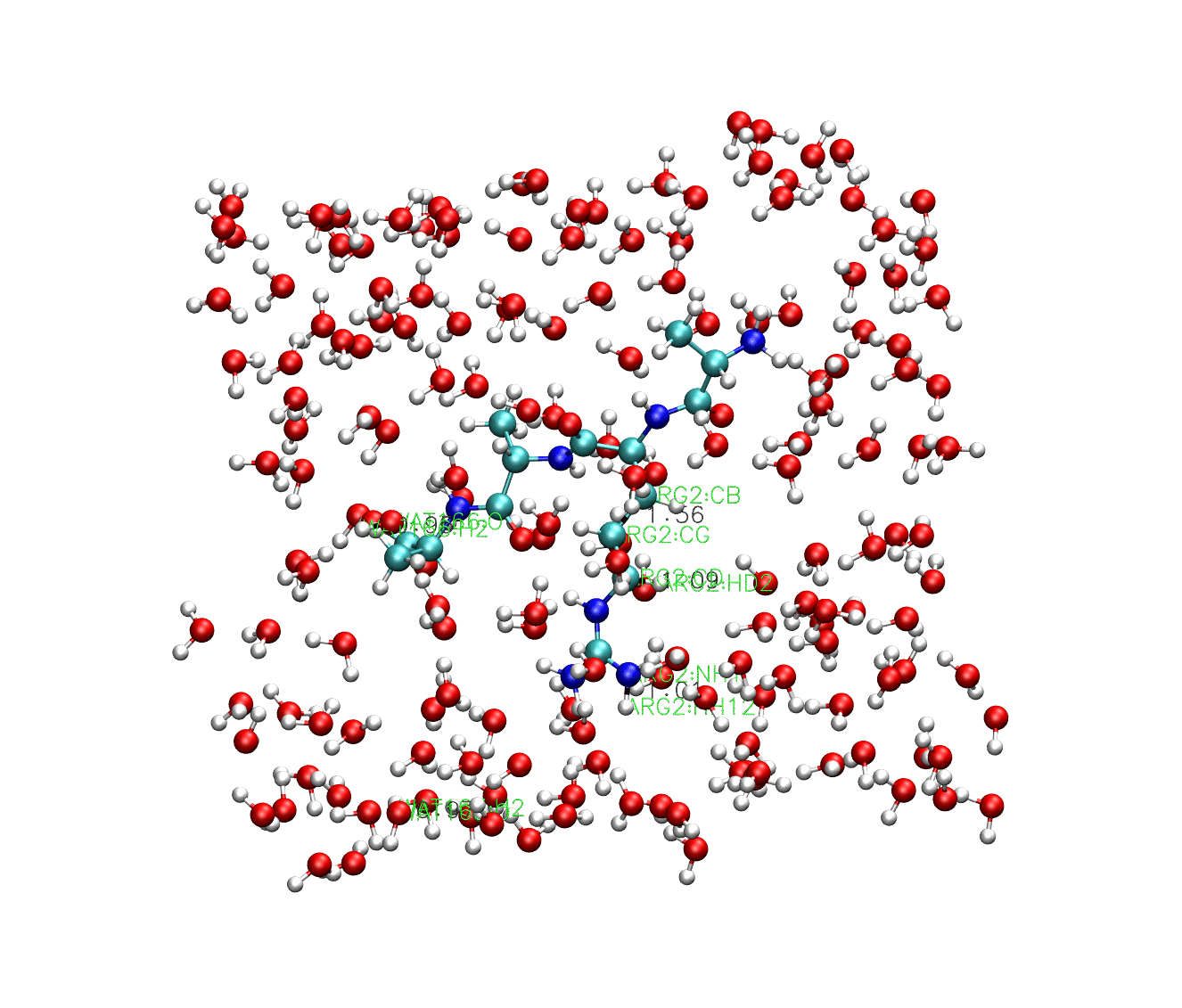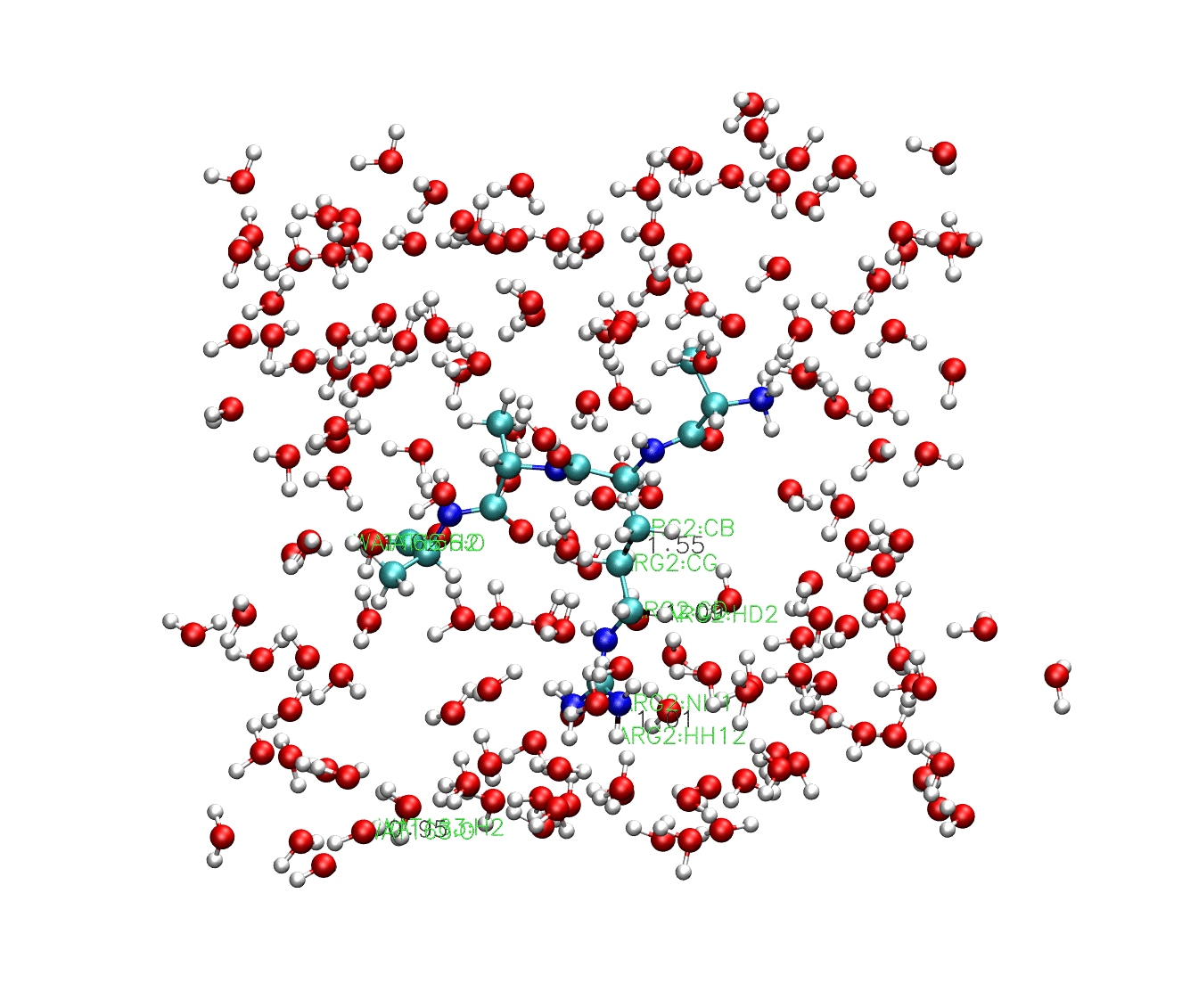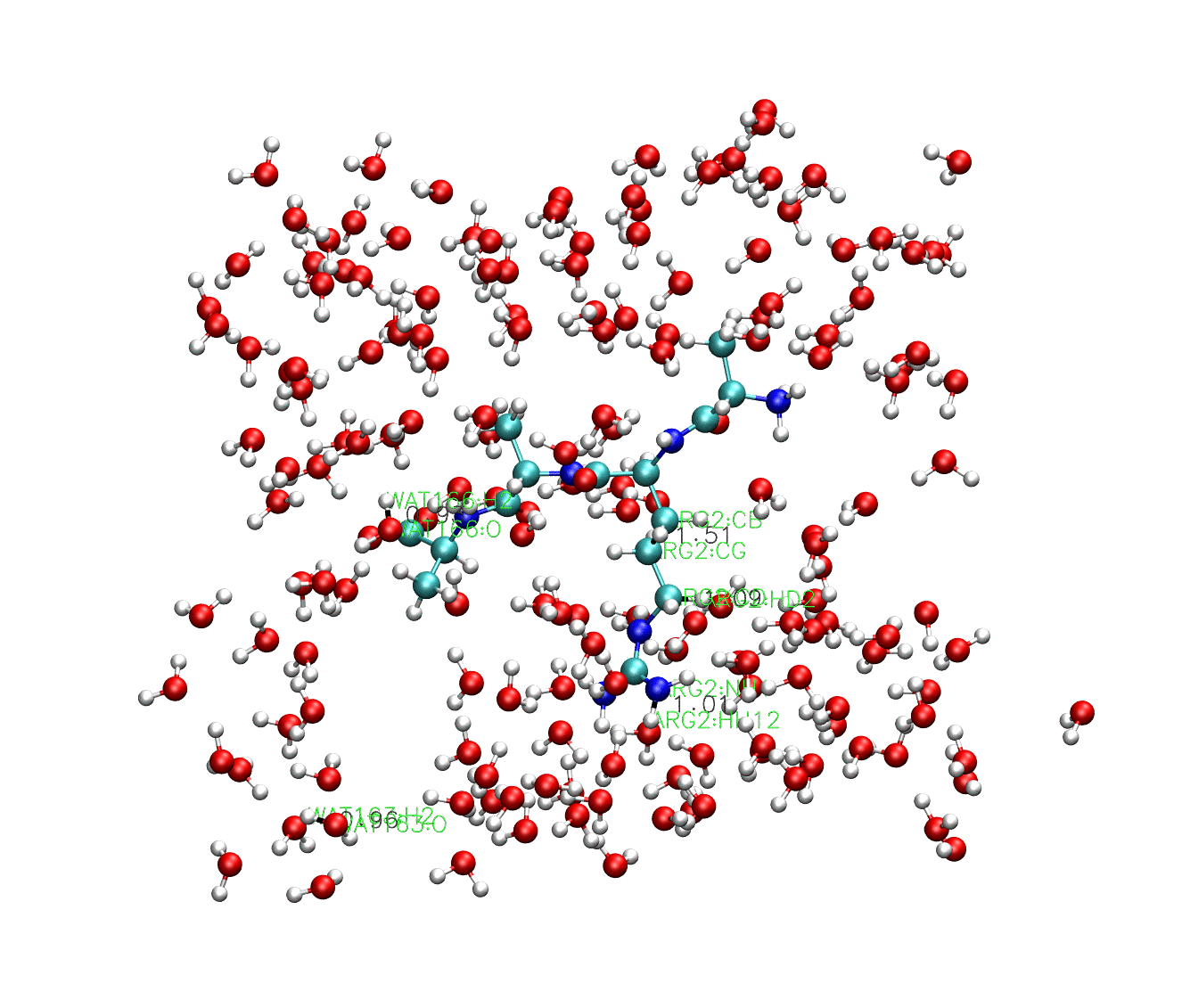
從這個結果中我們可以看到,因為沒有受到約束,水分子的鍵長鍵角都在不停的變化,多肽中的重原子共價鍵也在變化,只有多肽中的氫鍵長度保持不變。當然,如果這個案例中把程式碼調整為:
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin',
constraint=Lincs(system, 'all-bonds'))
那麼就能夠約束多肽裡面所有的鍵,而水分子不進行約束。
全域性氫鍵約束
上一個章節中提到了對多肽中的氫鍵用Lincs演演算法進行約束,再往上一個章節介紹了用SETTLE演演算法約束水分子,那麼這裡我們介紹如何混合使用兩種不同的約束演演算法。還是同樣的這個混合體系,我們在UpdaterMD中設定constraint的時候,不指定具體的Constraint約束演演算法,直接傳入一個h-bonds,那麼就會對全域性的氫鍵(共價鍵)進行約束。這時候會出現一個問題,就是水分子和多肽分子中都存在氫鍵,那麼哪些是使用SETTLE約束,哪些是使用Lincs約束的呢?有一個比較簡單的等級區分,SETTLE約束演演算法的優先順序要高於Lincs演演算法。所以,如果template組態檔中包含有SETTLE約束演演算法,那麼在設定相應原子的約束條件的時候就會優先使用SETTLE演演算法。所以在這個案例中,水分子部分會被SETTLE演演算法約束,而多肽部分的氫鍵會被Lincs演演算法約束。
from mindspore import nn, context
from sponge import ForceField, Sponge, set_global_units, Protein, UpdaterMD
from sponge.function import VelocityGenerator
from sponge.callback import RunInfo, WriteH5MD
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
set_global_units('A', 'kcal/mol')
system = Protein('../pdb/case1.pdb', template=['protein0.yaml'], rebuild_hydrogen=True)
system.fill_water(edge=4, template='water.tip3p.yaml')
energy = ForceField(system, parameters=['AMBER.FF99SB', 'TIP3P'], use_pme=False)
min_opt = nn.Adam(system.trainable_params(), 1e-03)
md = Sponge(system, potential=energy, optimizer=min_opt)
run_info = RunInfo(200)
md.run(2000, callbacks=[run_info])
# 定義一個新的迭代器,並用change_optimizer完成切換
vgen = VelocityGenerator(300)
velocity = vgen(system.shape, system.atom_mass)
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin',
constraint='h-bonds')
md.change_optimizer(opt)
nvt_h5md = WriteH5MD(system, 'test_nvt.h5md', save_freq=10, write_image=False)
md.run(2000, callbacks=[run_info, nvt_h5md])
程式碼執行結果如下:
[MindSPONGE] Adding 45 hydrogen atoms for the protein molecule in 0.002 seconds.
[MindSPONGE] The box size used when filling water is: [21.184929 22.856928 15.495929]
[MindSPONGE] The edge gap along x axis is 4.0.
[MindSPONGE] The edge gap along y axis is 3.999999523162842.
[MindSPONGE] The edge gap along z axis is 3.999999761581421.
[MindSPONGE] Totally 172 waters is added to the system!
[MindSPONGE] Adding water molecule task finished!
[MindSPONGE] Started simulation at 2023-09-05 17:42:44
[MindSPONGE] Step: 0, E_pot: 6019.2305
[MindSPONGE] Step: 200, E_pot: 3667.4216
[MindSPONGE] Step: 400, E_pot: 3007.9355
[MindSPONGE] Step: 600, E_pot: 2633.8372
[MindSPONGE] Step: 800, E_pot: 2380.7073
[MindSPONGE] Step: 1000, E_pot: 2233.6892
[MindSPONGE] Step: 1200, E_pot: 2129.8655
[MindSPONGE] Step: 1400, E_pot: 2050.8716
[MindSPONGE] Step: 1600, E_pot: 1993.4336
[MindSPONGE] Step: 1800, E_pot: 1949.2134
[MindSPONGE] Finished simulation at 2023-09-05 17:43:30
[MindSPONGE] Simulation time: 46.60 seconds.
--------------------------------------------------------------------------------
[MindSPONGE] The settle constraint is used for the molecule system.
[MindSPONGE] The lincs constraint is used for the molecule system.
[MindSPONGE] Started simulation at 2023-09-05 17:43:31
[MindSPONGE] Step: 0, E_pot: 1910.4988, E_kin: 514.44617, E_tot: 2424.9448, Temperature: 301.7242, Pressure: -1060987.0, Volume: 7503.4756
[MindSPONGE] Step: 200, E_pot: 2022.5985, E_kin: 230.11551, E_tot: 2252.714, Temperature: 197.94637, Pressure: -13623341.0, Volume: 7503.4756
[MindSPONGE] Step: 400, E_pot: 1955.0367, E_kin: 234.73169, E_tot: 2189.7686, Temperature: 201.91722, Pressure: -14059788.0, Volume: 7503.4756
[MindSPONGE] Step: 600, E_pot: 1940.77, E_kin: 225.15894, E_tot: 2165.929, Temperature: 193.6827, Pressure: -13967141.0, Volume: 7503.4756
[MindSPONGE] Step: 800, E_pot: 1921.0624, E_kin: 215.5067, E_tot: 2136.569, Temperature: 185.3798, Pressure: -14294088.0, Volume: 7503.4756
[MindSPONGE] Step: 1000, E_pot: 1901.498, E_kin: 221.8313, E_tot: 2123.3293, Temperature: 190.82025, Pressure: -14622425.0, Volume: 7503.4756
[MindSPONGE] Step: 1200, E_pot: 1894.645, E_kin: 206.15628, E_tot: 2100.8013, Temperature: 177.33653, Pressure: -14258592.0, Volume: 7503.4756
[MindSPONGE] Step: 1400, E_pot: 1878.4142, E_kin: 205.46025, E_tot: 2083.8745, Temperature: 176.73781, Pressure: -14435189.0, Volume: 7503.4756
[MindSPONGE] Step: 1600, E_pot: 1877.7242, E_kin: 209.3739, E_tot: 2087.0981, Temperature: 180.10434, Pressure: -14059375.0, Volume: 7503.4756
[MindSPONGE] Step: 1800, E_pot: 1865.3273, E_kin: 215.8046, E_tot: 2081.1318, Temperature: 185.63606, Pressure: -14290035.0, Volume: 7503.4756
[MindSPONGE] Finished simulation at 2023-09-05 17:45:46
[MindSPONGE] Simulation time: 2 minutes 14.8 seconds.
--------------------------------------------------------------------------------
可以看到,在視覺化的結果中,水分子的鍵長鍵角是保持不動的,多肽中的氫鍵長度是保持不動的,但是多肽中的重原子共價鍵因為不受約束,所以也一直在動。
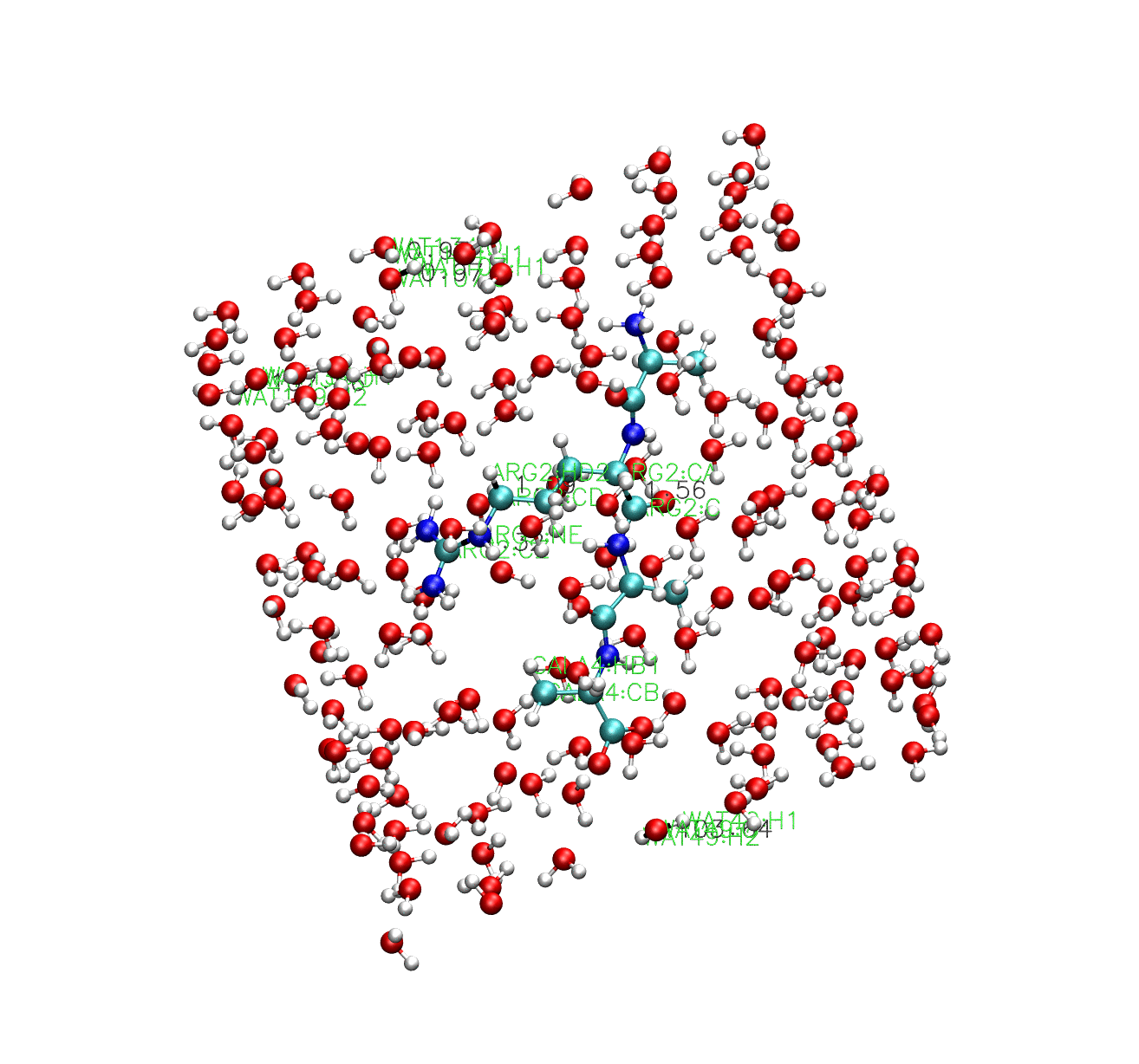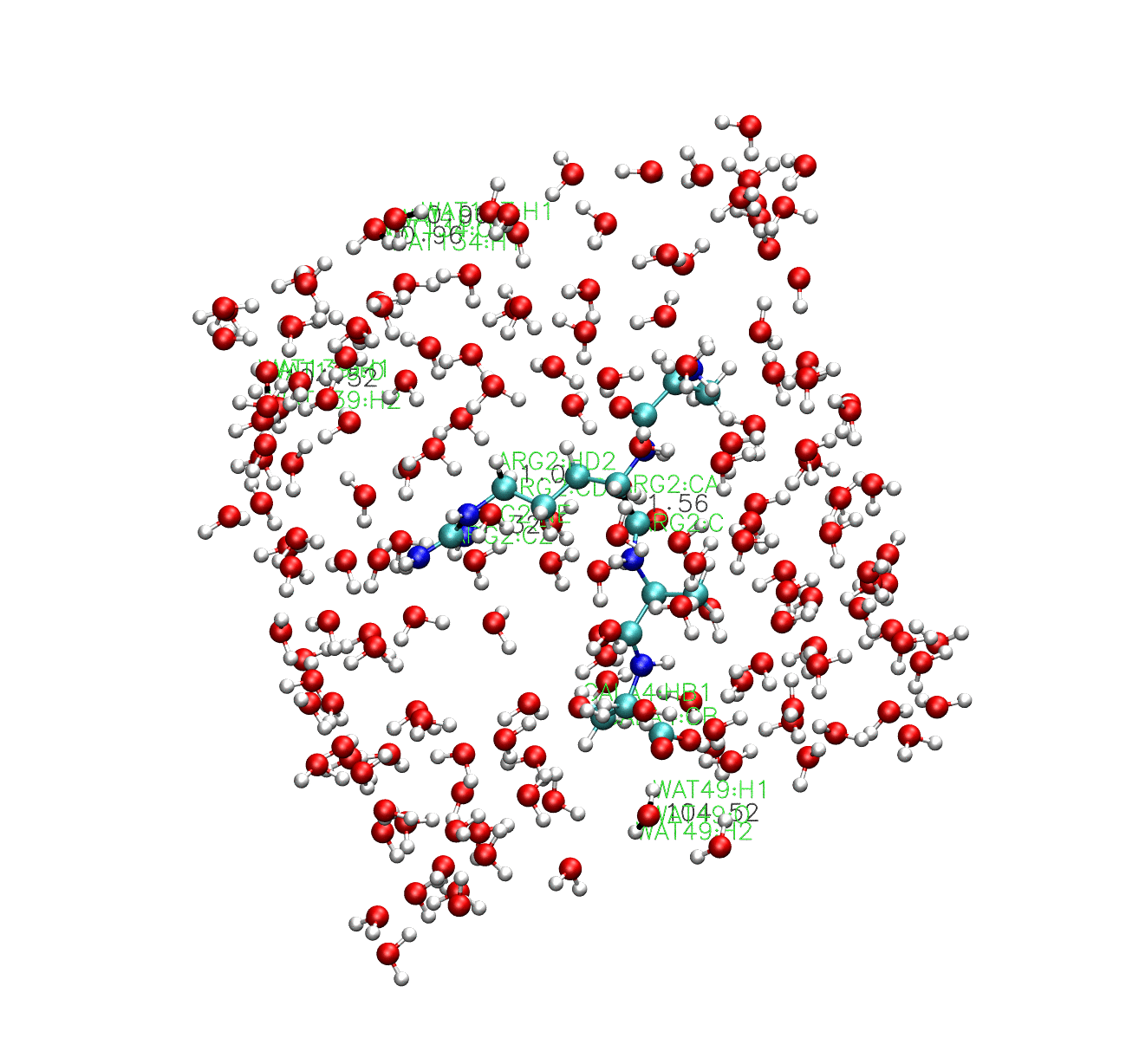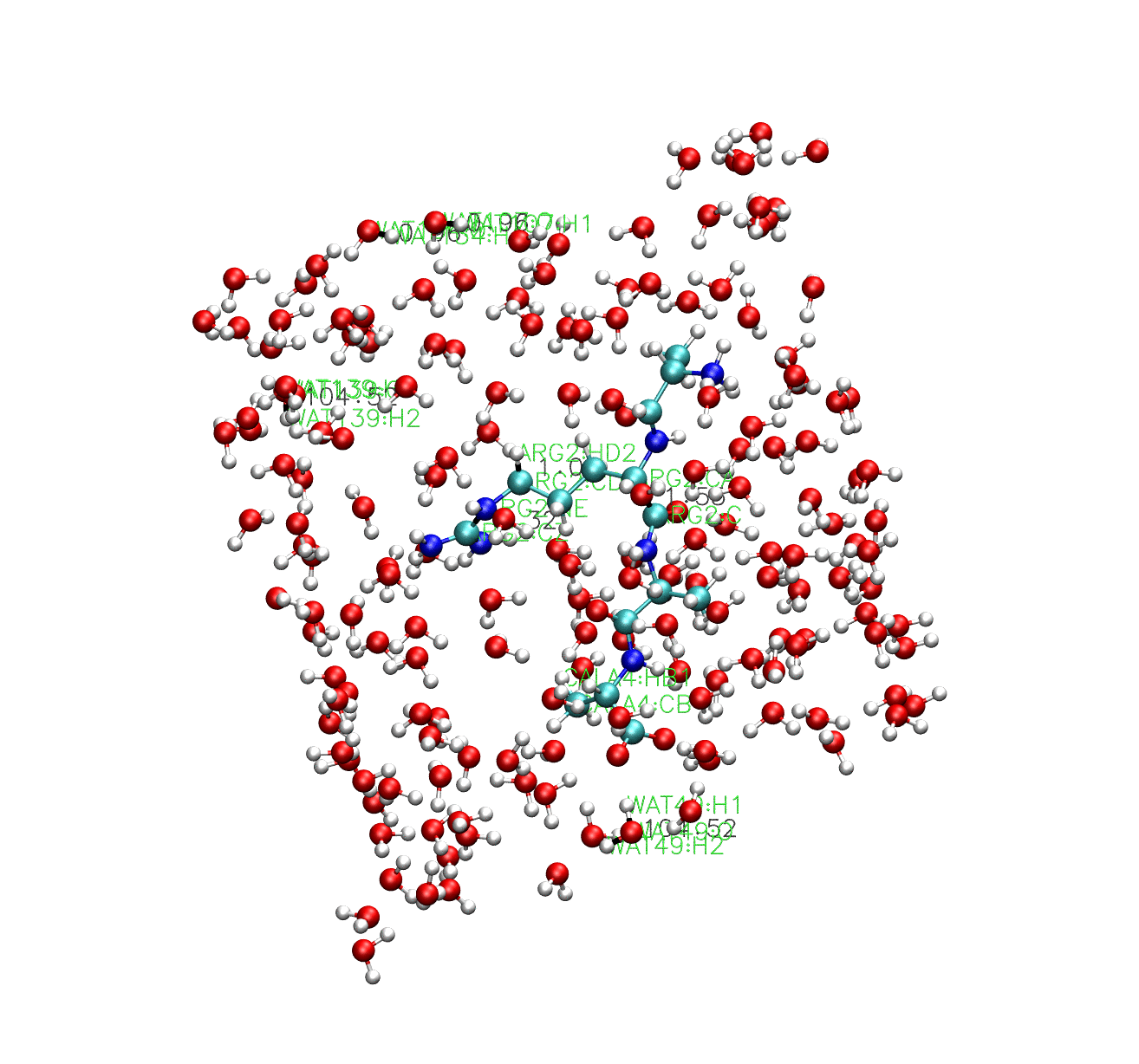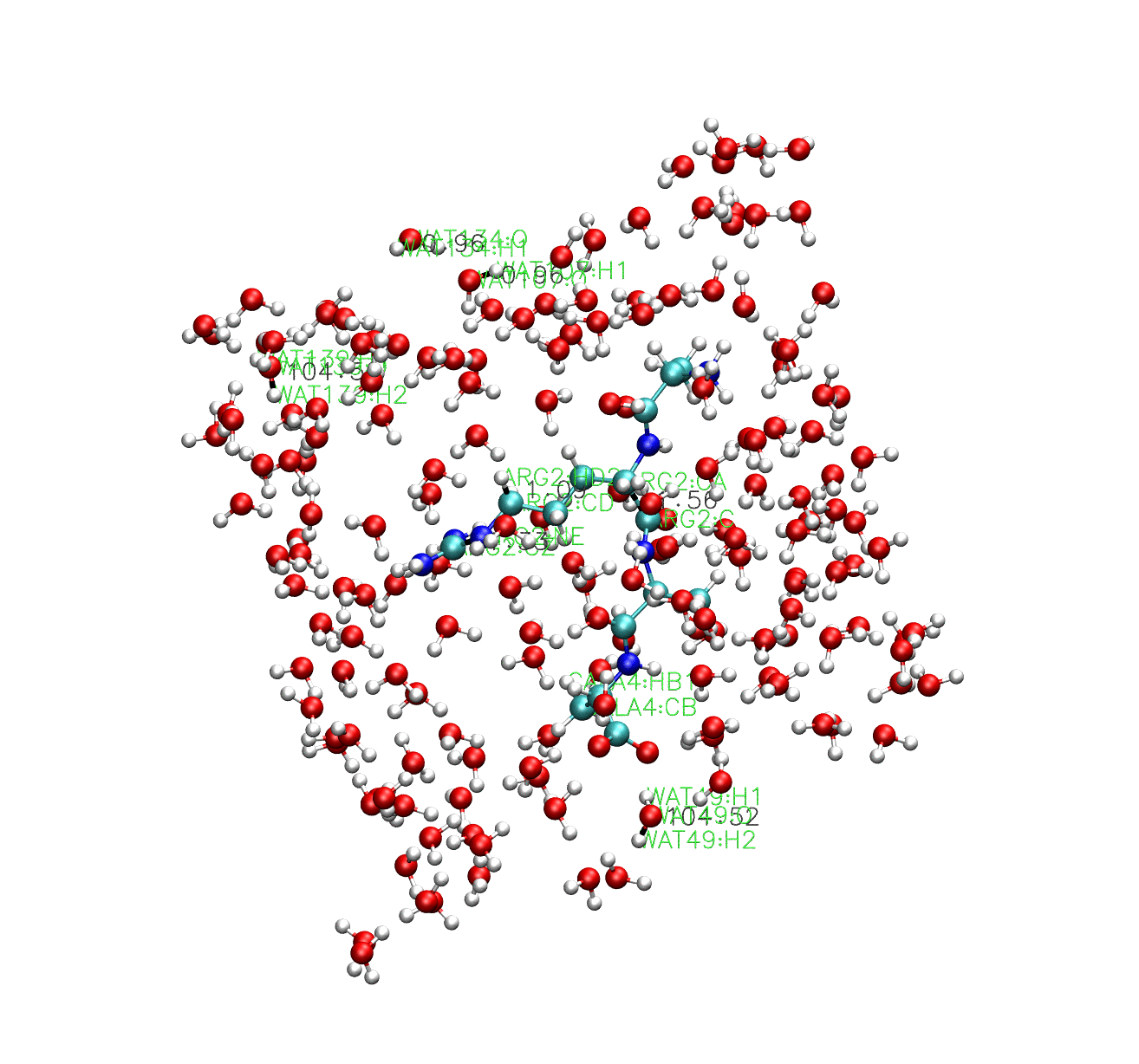
假如我們希望用Lincs演演算法來約束水分子中的氫鍵,那麼可以在匯入模板的時候,匯入一個沒有SETTLE引數的template,這樣就可以實現用Lincs去約束水分子。但是需要注意的是,一個水分子只有兩根共價鍵,因此Lincs演演算法雖然可以約束水分子的兩個氫鍵的鍵長,但是無法約束其鍵角引數,也因此不能與TIP4P模型配合使用。
全體共價鍵約束
跟上面一個案例比較類似的,上一個案例是約束整個體系中所有的氫鍵,這裡直接約束所有的共價鍵。只需要把h-bonds改為all-bonds即可。
from mindspore import nn, context
from sponge import ForceField, Sponge, set_global_units, Protein, UpdaterMD
from sponge.function import VelocityGenerator
from sponge.callback import RunInfo, WriteH5MD
context.set_context(mode=context.GRAPH_MODE, device_target='GPU', device_id=1)
set_global_units('A', 'kcal/mol')
system = Protein('../pdb/case1.pdb', template=['protein0.yaml'], rebuild_hydrogen=True)
system.fill_water(edge=4, template='water.tip3p.yaml')
energy = ForceField(system, parameters=['AMBER.FF99SB', 'TIP3P'], use_pme=False)
min_opt = nn.Adam(system.trainable_params(), 1e-03)
md = Sponge(system, potential=energy, optimizer=min_opt)
run_info = RunInfo(200)
md.run(2000, callbacks=[run_info])
# 定義一個新的迭代器,並用change_optimizer完成切換
vgen = VelocityGenerator(300)
velocity = vgen(system.shape, system.atom_mass)
opt = UpdaterMD(system=system,
time_step=1e-3,
velocity=velocity,
integrator='velocity_verlet',
temperature=300,
thermostat='langevin',
constraint='all-bonds')
md.change_optimizer(opt)
nvt_h5md = WriteH5MD(system, 'test_nvt.h5md', save_freq=10, write_image=False)
md.run(2000, callbacks=[run_info, nvt_h5md])
執行結果如下:
[MindSPONGE] Adding 45 hydrogen atoms for the protein molecule in 0.003 seconds.
[MindSPONGE] The box size used when filling water is: [21.184929 22.856928 15.495929]
[MindSPONGE] The edge gap along x axis is 4.0.
[MindSPONGE] The edge gap along y axis is 3.999999523162842.
[MindSPONGE] The edge gap along z axis is 3.999999761581421.
[MindSPONGE] Totally 172 waters is added to the system!
[MindSPONGE] Adding water molecule task finished!
[MindSPONGE] Started simulation at 2023-09-06 09:10:50
[MindSPONGE] Step: 0, E_pot: 6019.2305
[MindSPONGE] Step: 200, E_pot: 3667.4207
[MindSPONGE] Step: 400, E_pot: 3007.9258
[MindSPONGE] Step: 600, E_pot: 2633.8313
[MindSPONGE] Step: 800, E_pot: 2380.703
[MindSPONGE] Step: 1000, E_pot: 2233.685
[MindSPONGE] Step: 1200, E_pot: 2129.8577
[MindSPONGE] Step: 1400, E_pot: 2050.8628
[MindSPONGE] Step: 1600, E_pot: 1993.4271
[MindSPONGE] Step: 1800, E_pot: 1949.2087
[MindSPONGE] Finished simulation at 2023-09-06 09:11:36
[MindSPONGE] Simulation time: 45.36 seconds.
--------------------------------------------------------------------------------
[MindSPONGE] The settle constraint is used for the molecule system.
[MindSPONGE] The lincs constraint is used for the molecule system.
[MindSPONGE] Started simulation at 2023-09-06 09:11:37
[MindSPONGE] Step: 0, E_pot: 1910.496, E_kin: 491.1751, E_tot: 2401.6711, Temperature: 288.07568, Pressure: -1245873.8, Volume: 7503.4756
[MindSPONGE] Step: 200, E_pot: 2011.3496, E_kin: 225.65253, E_tot: 2237.0022, Temperature: 198.51881, Pressure: -13732667.0, Volume: 7503.4756
[MindSPONGE] Step: 400, E_pot: 1960.8837, E_kin: 220.33536, E_tot: 2181.219, Temperature: 193.841, Pressure: -14297195.0, Volume: 7503.4756
[MindSPONGE] Step: 600, E_pot: 1923.8737, E_kin: 220.88599, E_tot: 2144.7598, Temperature: 194.32542, Pressure: -13654464.0, Volume: 7503.4756
[MindSPONGE] Step: 800, E_pot: 1913.1719, E_kin: 193.6853, E_tot: 2106.8572, Temperature: 170.39551, Pressure: -14154988.0, Volume: 7503.4756
[MindSPONGE] Step: 1000, E_pot: 1888.8213, E_kin: 197.11978, E_tot: 2085.9412, Temperature: 173.417, Pressure: -13763211.0, Volume: 7503.4756
[MindSPONGE] Step: 1200, E_pot: 1882.9513, E_kin: 208.48392, E_tot: 2091.4353, Temperature: 183.41466, Pressure: -14133313.0, Volume: 7503.4756
[MindSPONGE] Step: 1400, E_pot: 1861.6063, E_kin: 197.9073, E_tot: 2059.5137, Temperature: 174.10983, Pressure: -14121973.0, Volume: 7503.4756
[MindSPONGE] Step: 1600, E_pot: 1850.9318, E_kin: 218.16049, E_tot: 2069.0923, Temperature: 191.92766, Pressure: -14353631.0, Volume: 7503.4756
[MindSPONGE] Step: 1800, E_pot: 1856.4893, E_kin: 205.427, E_tot: 2061.9163, Temperature: 180.72531, Pressure: -14140336.0, Volume: 7503.4756
[MindSPONGE] Finished simulation at 2023-09-06 09:13:51
[MindSPONGE] Simulation time: 2 minutes 14.2 seconds.
--------------------------------------------------------------------------------
在最後這個視覺化結果中我們可以看到,所有的共價鍵的長度都被固定住了。不過這裡面的水分子還是使用了SETTLE約束而不是Lincs約束。從列印的輸出結果我們也可以看到,兩個約束演演算法都有被呼叫到。
總結概要
本文主要介紹了在MindSponge中使用SETTLE和Lincs約束演演算法的方法,以及相關演演算法的簡單原理。SETTLE約束演演算法主要應用於水分子體系,限制的是一個等腰三角形的拓撲結構,特點是可並行,效能較好。Lincs約束演演算法更多的被應用在蛋白質體系,主要限制的是每一個共價鍵的鍵長,特點是適用體系比較靈活,但總體計算量較大,且不可並行化。
版權宣告
本文首發連結為:https://www.cnblogs.com/dechinphy/p/constraint.html
作者ID:DechinPhy
更多原著文章:https://www.cnblogs.com/dechinphy/
請博主喝咖啡:https://www.cnblogs.com/dechinphy/gallery/image/379634.html