MindSponge分子動力學模擬——軟體架構(2023.08)
技術背景
在前面一篇文章中,我們介紹了MindSponge的兩種不同的安裝與使用方法,讓大家能夠上手使用。這篇文章主要講解MindSponge的軟體架構,並且協同mindscience倉庫講解一下二者的區別。
整體架構
首先我們來了解一下MindSponge獨立倉庫的軟體架構,其實核心部分的軟體架構跟mindscience是一致的。
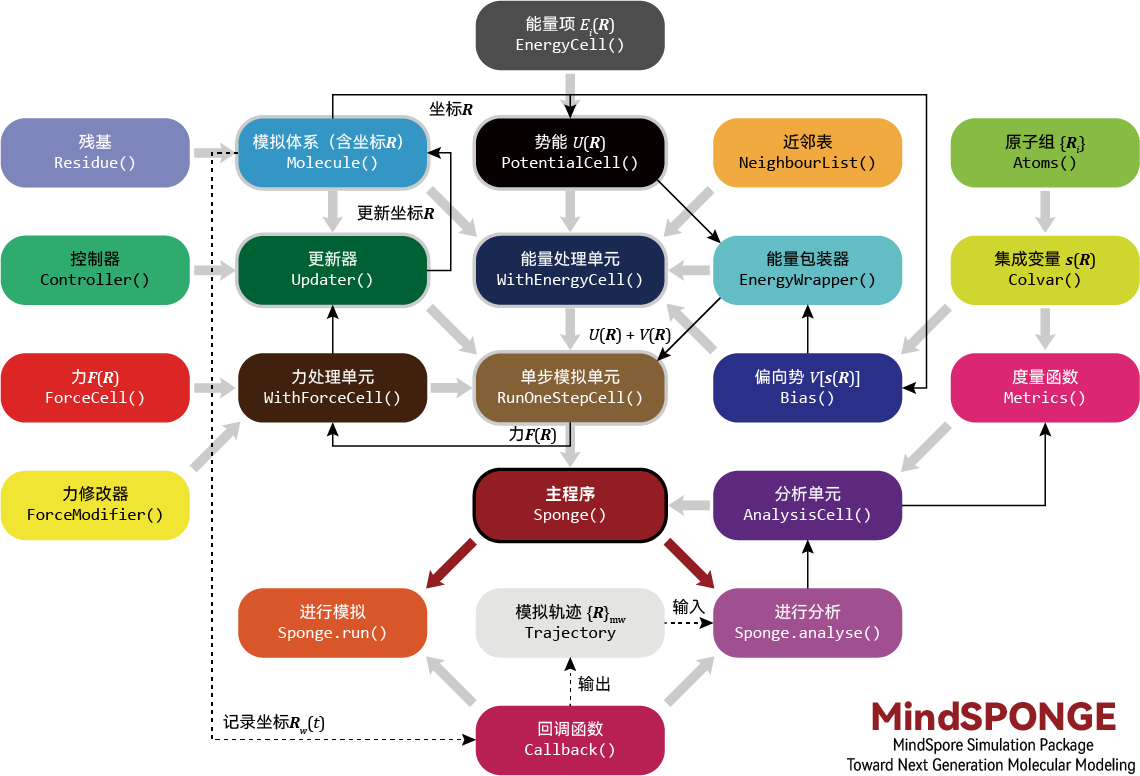
在這個架構圖中,我們不僅可以看到MindSponge的內部模組劃分,還能看到一個分子模擬資料處理的流程。
-
首先從一個模擬體系Molecule()開始,這個Molecule()可以獨立定義,可以自行封裝,也可以用Residue()來構建,裡面儲存有分子的基本資訊,如座標、原子名稱等。
-
然後根據Molecule()提供的資訊構建一個力場,形成一個PotentialCell()勢能函數。這個勢能函數,是基於模組化的EnergyCell()搭建的一個整體。而且除了力場本身之外,還可以接收外界輸入的EnergyWrapper(),可用於新增神經網路力場,或者是增強取樣產生的Bias()。這也是該架構的一個先進性的體現,雖然是一個MD軟體,但不僅僅侷限於做MD。
-
我們可以使用MindSpore內建的優化器,如Adam等,對Molecule()的Parameter進行更新迭代,可以自定義Updater()來對Molecule()進行更新。一般情況下,更新的依據主要來自於對PotentialCell()的自動微分。當然,也可以自行定義ForceCell()的內容。如果我們在動力學模擬的過程中,需要定義一些約束演演算法,或者是控溫控壓演演算法,都可以將相關的Controller()傳入到Updater()中。
-
接下來的重點是要通過PotentialCell()來獲取力,如果是以往傳統的做法,只能通過取兩點做差分的方法來得到一個作用力。但如果我們這裡的所有計算都通過MindSpore的內建運算元來實現的話,就可以使用MindSpore的自動微分來計算這個力。最終我們會得到一個ForceCell()傳到Updater()裡面,但是這一步對使用者是不感知的,使用者只需要定義好PotentialCell()這一塊就足夠了。或者使用者也可以自行定義一個ForceModifier()傳入到WithForceCell(),來構建一個自定義力場。
-
在具備了體系Molecule()、優化器Optimizer()和力場WithForceCell()之後,我們就可以開始基本的動力學模擬計算,此時就需要用到主程式Sponge()來對整個流程進行管理。並且,我們可以定義一些回撥函數CallBack()給Sponge()進行任務追蹤。比如RunInfo()可以在螢幕上輸出指定步長的能量,或者是WriteH5MD可以將整個MD的軌跡儲存到一個指定的hdf5格式的檔案中,檔案字尾名為
h5md,可以在VMD中增加一個hdf5的外掛來進行動態視覺化。
軟體模組
我們先來看一下MindSponge這邊的軟體專案主頁:
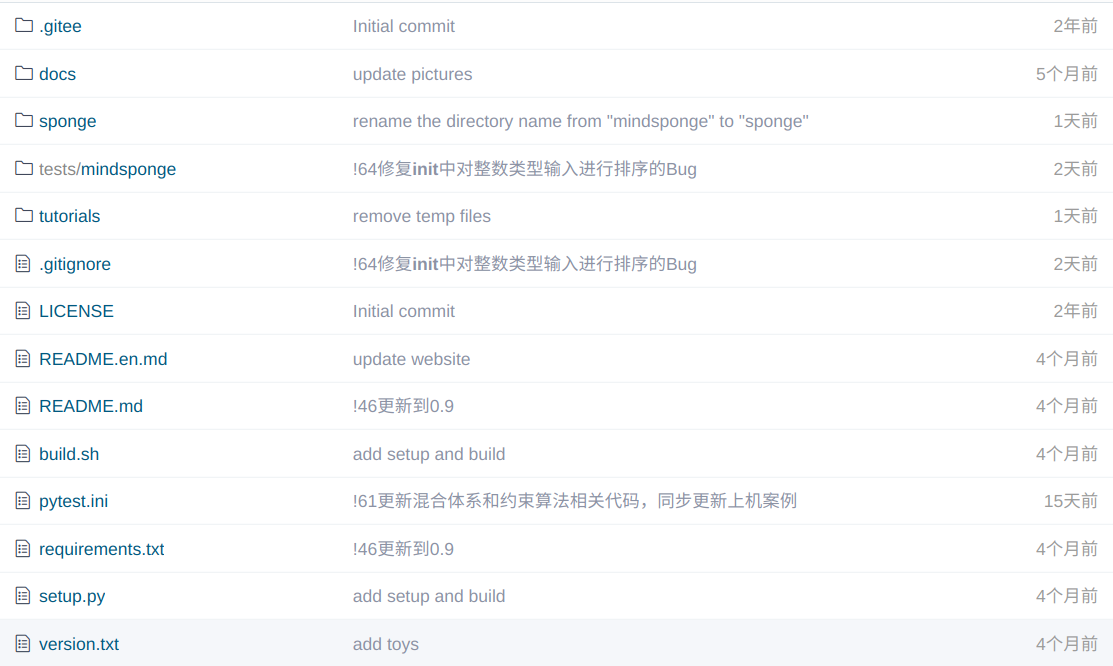
針對於這其中的內容我們簡單梳理一下:sponge/是核心目錄,tutorials/和tests/顯然是一些案例或者是測試用例的路徑,docs/是一些檔案或者是圖片,其他的檔案基本上是一些跟mindsponge倉庫的安裝相關的內容,所以我們重點關注下sponge/下的內容:
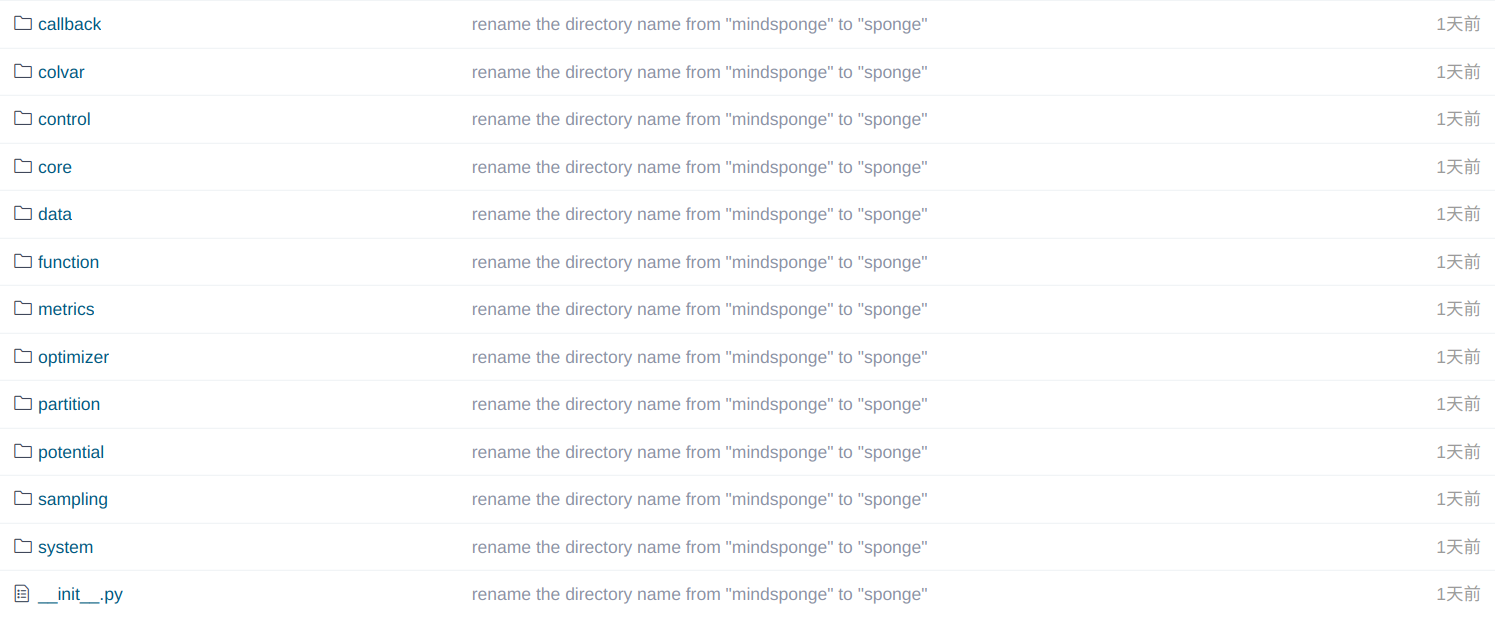
這裡我們對照每一個目錄來進行內容解析:
-
callback:回撥函數。在執行分子動力學模擬程式的過程中,我們可能會有記錄一些能量、力、速度、軌跡的需求,這時候就需要呼叫回撥函數,對相應的內容進行輸出。目前比較常用的回撥函數,是RunInfo和WriteH5MD。RunInfo可以在螢幕上輸出執行的結果,WriteH5MD則是把軌跡等輸出到一個hdf5格式的檔案裡面,字尾為
*.h5md,可以用https://gitee.com/helloyesterday/VMD-h5mdplugin這個VMD外掛來進行視覺化。 -
colvar:各種形式的參量。這裡預定義了一些常用的參量,比如分子質心、原子間鍵長鍵角等。當然,使用者也可以自己開發一些參量,可以用於增強取樣。
-
control:控制器和約束演演算法。顧名思義,就是要對原子系統迭代的過程進行控制,比如溫度引數和壓強引數,甚至是控制鍵長鍵角,都是可以的。
-
core:主程式。這裡就是Sponge()的存放路徑,對整個模擬過程進行管理。
-
data:引數檔案和模板檔案。我們在使用模板構建分子系統力場的時候,會使用到一些模板檔案和力場引數檔案,這些檔案就都儲存在data目錄下,並且有相應的檔案讀取函數。
-
function:非內建函數。對於一些公用的函數,一般都集中放在function路徑下。
-
metrics:度量函數。在機器學習中一般該函數被用於衡量模型的好壞,這裡我們一般就用來計算某個特定的引數,比如設定一個自定義的CV函數,可以與colvar中的內容配合使用。
-
optimizer:優化器和積分器。之所以我們可以使用AI框架來實現一個分子動力學模擬的框架,正是得益於分子動力學模擬與AI訓練/推理之中的共性。在神經網路的訓練中我們可以使用優化器來迭代損失函數,而在分子動力學模擬中就可以使用積分器(如Leap-Frog和Velocity-Verlet)來迭代勢能函數。
-
partition:近鄰表。在分子系統較大時,就不能考慮全連線的相互作用,只能考慮區域性相互作用。而分子模擬的過程中,近鄰表實際上每一步都在變化,因此需要一個單獨用於計算近鄰關係的Cell。
-
potential:勢能函數。這個就不需要過多解釋了,相當於力場裡面每一項的內容分開寫在了幾個檔案裡面。
-
sampling:增強取樣函數。可用於修改勢能項,也可以直接修改力,可以加快取樣的程序。
-
system:分子系統基礎類別。儲存有一個分子系統的基本資訊,如原子名稱、殘基名稱,還有最核心的原子構象座標等等。
總結概要
分子模擬具有眾多的應用場景,比如製藥領域和材料領域,做好分子模擬的工作,可以極大程度上縮減新葯物新材料的研發成本和研發週期。近幾年隨著GPT-4和Diffusion Model的大火,讓大家意識到了AI已經具備了相當的解決問題的能力。因此基於AI的框架和模型,對比AI訓練與分子模擬之間的共性,可以實現一個面向AI時代的分子模擬框架。本文主要介紹基於MindSpore框架實現的,MindSponge分子動力學模擬框架的軟體架構。
版權宣告
本文首發連結為:https://www.cnblogs.com/dechinphy/p/structure.html
作者ID:DechinPhy
更多原著文章請參考:https://www.cnblogs.com/dechinphy/
打賞專用連結:https://www.cnblogs.com/dechinphy/gallery/image/379634.html
騰訊雲專欄同步:https://cloud.tencent.com/developer/column/91958
CSDN同步連結:https://blog.csdn.net/baidu_37157624?spm=1008.2028.3001.5343
51CTO同步連結:https://blog.51cto.com/u_15561675