【論文筆記】UNet
語意分割的U-Net網路結構Unet是2015年誕生的模型,它幾乎是當前segmentation專案中應用最廣的模型。Unet能從更少的訓練影象中進行學習,當它在少於40張圖的生物醫學資料集上訓練時,IOU值仍能達到92%。Unet網路非常簡單,前半部分作用是特徵提取,後半部分是上取樣。在一些文獻中也把這樣的結構叫做編碼器-解碼器結構。由於此網路整體結構類似於大寫的英文字母U,故得名U-net。
論文連結: https://arxiv.org/pdf/1505.04597v1.pdf
github: https://github.com/milesial/Pytorch-UNet
1 Motivation
生物醫學影象處理面臨的問題
- 經典折積網路大部分都是針對影象分類任務的,但是在一些特定場景,如醫療影象處理領域,應是pixel-wise畫素級的處理,輸入輸出均是影象,即影象分割。
- 生物醫學任務中沒有很多標註的資料集
為了解決這兩個問題,Ciresan用滑窗法來預測patch的類別(patch指畫素周圍的區域性區域)。 該演演算法有兩個主要問題:(1)由於每個patch都需要訓練導致這個演演算法很慢,且patch之間有很多重複。(2)定位準確率和上下文聯絡之間需要平衡,patch越大需要pooling越多準確率越低,patch越小則不具備上下文聯絡。
2 U-Net網路
作者以FCN全折積神經網路為基礎設計了Unet,其中包含兩條串聯的路徑:contracting path用來提取影象特徵,捕捉context,將影象壓縮為由特徵組成的feature maps;expanding path用來精準定位,將提取的特徵解碼為與原始影象尺寸一樣的分割後的預測影象。
和FCN相比,U-Net的第一個特點是完全對稱,也就是左邊和右邊是很類似的,而FCN的decoder相對簡單,只用了一個deconvolution的操作,之後並沒有跟上折積結構。第二個區別就是skip connection,FCN用的是加操作(sum),U-Net用的是疊操作(concat)。最重要的是編碼和解碼(encoder-decoder)的思路,編碼和解碼常用於壓縮影象和去噪聲,後來這個思路被用在了影象分割上,非常簡潔好用。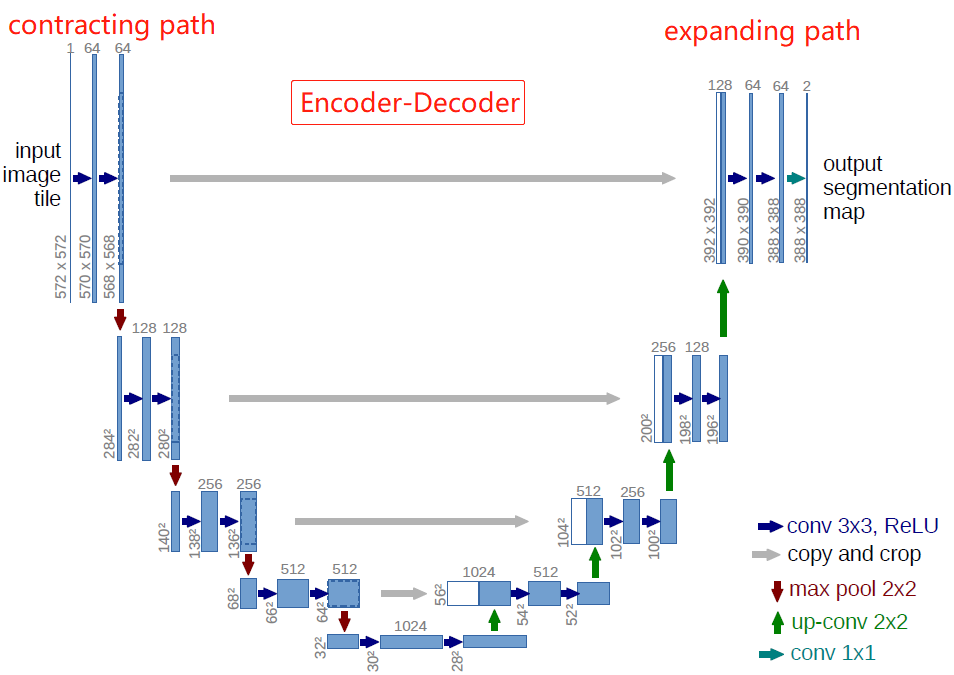
- 網路左邊一側作者稱之為contracting path,右邊一側為expanding path。
- 藍色箭頭為折積層,折積層的stride=1,padding=0,因此折積後特徵層的寬高會減2。折積層後接ReLU啟用函數,沒有BN層(BN由Google於2015年提出)。
- 池化層stride=2,池化後寬高減半,通道數不變。池化層之後的折積層將通道數翻倍。
- 綠色的up-conv是轉置折積,將特徵層的寬高×2,通道數減半。
- 灰色copy and crop是先對左邊的特徵層進行中心裁剪(保留中心特徵),再與右邊path對應的特徵層進行通道數上的concat。
- 最後的1×1的折積沒有ReLU,輸出通道數為類別數。
Overlap-tile
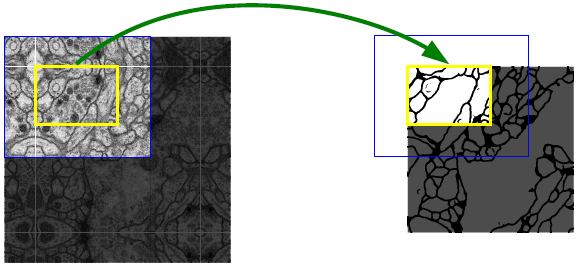
可以發現Unet論文中輸入的影象是572×572,但是輸出影象大小為388×388。也就是說推理上圖黃色部分,需要藍色區域內的影象資料作為輸入。當黃色區域位於邊緣時,就會產生邊緣資料缺失的情況(上圖右邊藍框中的空白部分)。我們可以在預處理中,對輸入影象進行padding,通過padding擴大輸入影象的尺寸,使得最後輸出的結果正好是原始影象的尺寸,同時輸入影象塊(黃框)的邊界也獲得了上下文資訊從而提高預測的精度,本文用的是mirror padding。我們自己搭建網路的時候,輸入輸出往往是一樣大小的(padding=1),因此不需要考慮這個問題。
3 訓練
3.1 資料增強
網路需要大量標註訓練樣本,生物醫學任務中沒有數千個標註的資料集,所以需要對資料進行資料擴張。作者採用了彈性變形的影象增廣,以此讓網路學習更穩定的影象特徵。因為資料集是細胞組織的影象,細胞組織的邊界每時每刻都會發生不規則的畸變,所以這種彈性變形的增廣是非常有效的。論文筆記:影象資料增強之彈性形變(Elastic Distortions)
3.2 損失函數的權重
細胞組織影象的一大特點是,多個同類的細胞會緊緊貼合在一起,其中只有細胞壁或膜組織分割。因此,作者在計算損失的過程中,給兩個細胞的邊緣部分及細胞間的背景部分增加了損失的權重,以此讓網路更加註重這類重合的邊緣資訊。
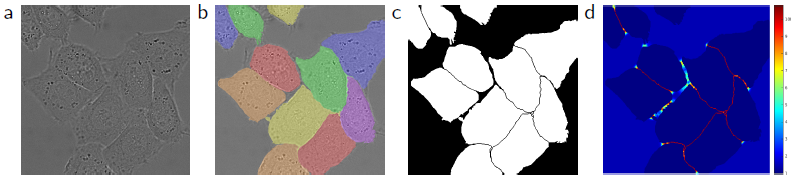
如上圖所示,圖(a)為原始影象,圖(b)為人工標註的範例分割ground truth,圖(c)為mask,圖(d)為每個畫素的損失權重weight map。首先用形態學操作獲得邊界,再用下面的公式計算weight map
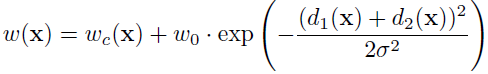
其中,wc是為了類別平衡,d1是該畫素到最近細胞邊界的距離,d2是到第二近的細胞邊界的距離。在作者實驗中設定w0=10,σ≈5pixels.
3.3 其他
- 優化器:SGD + momentum(0.99)
- batch:為了最大限度的使用GPU視訊記憶體,比起輸入一個大的batch size,更傾向於大量輸入tiles,因此實驗batch size為1。
- 損失函數:pixel-wise softmax + cross_entropy
- 初始化高斯分佈權重:在具有許多折積層和通過網路的不同路徑的深度網路中,權重的良好初始化非常重要。 否則,網路的某些部分可能會進行過多的啟用,而其他部分則永遠不會起作用。 理想情況下,應調整初始權重,以使網路中的每個特徵圖都具有大約單位方差。作者用的高斯分佈的權重。
參考
1. 精讀論文U-Net
2. 論文筆記:影象資料增強之彈性形變(Elastic Distortions)
3. 研習U-Net