R資料分析:孟德爾隨機化實操
好多同學詢問孟德爾隨機化的問題,我再來嘗試著梳理一遍,希望對大家有所幫助,首先看下圖1分鐘,盯著看將下圖印在腦海中:
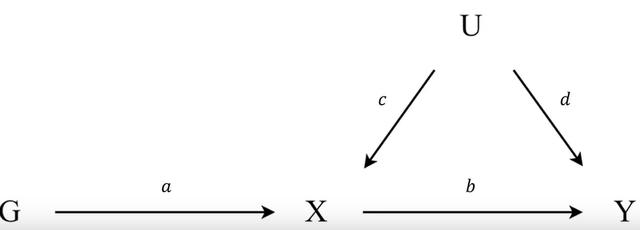
上圖是工具變數(不知道工具變數請翻之前的文章)的模式圖,明確一個點:我們做孟德爾的時候感興趣的是x和y的關係,也就是小b,但是我們直接去跑x對y的迴歸肯定是不對的,因為有很多的U,因此我們藉助工具變數G(關於工具變數我們之前的文章有詳細的解釋,請自行查閱),去估計我們感興趣的小b。
現在有天然良好的工具變數G,也就是我們的基因變數,此時有上面的圖,再次重申:我們感興趣的,最終希望得到準確估計的值是小b,按照上圖我們應該有GY的關係是ab,GX的關係是a,於是乎b可以寫成ab/a,就是我們感興趣的b可以換一種思路得到,如下:

上面的式子要跑通的話,我們需要知道G-Y的關係和G-X的關係。
但是我們GY也就是基因和結局的關係已經有人給我們研究好了,我們可以直接去GWAS裡面找研究好的summarydata拿來用就行。
但是我們的的GX也就是基因和暴露的關係也已經有人給我們研究好了,我們可以直接去GWAS裡面找研究好的summarydata拿來用就行。
也就是說,通過孟德爾隨機化,我們完全可以毫不費力地估計出我們需要的小b,也就是暴露和結局的關係----就是今天要再次給大家介紹的孟德爾隨機化研究。
思路就是這麼清晰。就是這麼清晰。搞不明白的同學再多讀幾遍。
術語解析
為了幫助大家理解思想,在孟德爾隨機化的實操中有幾個術語得提點一波:
連鎖不平衡(linkage disequilibrium):剛剛講我們可以有很多的基因結局/暴露的關係的,就是GWAS裡面好些基因可以用,這個時候我們不希望基因之間有相關(會造成double counting,使得結果偏倚):
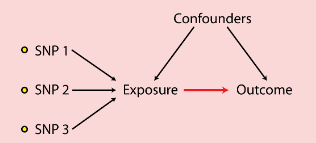
我們實際做的時候,模式是像上圖,snp之間你說不相干就不相干?當兩個位點的不同等位基因的關聯頻率高於或低於獨立隨機關聯的條件下的期望頻率,這種情況是客觀存在的,此時時這些工具變數之間相關性就叫連鎖不平衡,其大小可以用LD r方來表示,這個指標也是我們在操作時需要設定的指標之一。
水平基因多效性(Horizontal Pleiotropy):理解這個概念先看下圖:

意思是我的理想的情況是通過ab/a的操作估計出b,但是看上圖,是不是免不了會出現f這條路徑,如果出現了f,我們的基因和結局之間的關係就是f+ab,此時,我用原來的方法估計的就不是b了,而是b+f/a了,就不對了(始終記住我們關心的是b)。
但是如果我的基因變數很多,從而有很多的f,如果所有f的期望均值為0,那麼最後我們彙總一下得到的結果也基本上就是b了,無傷大雅。但是就怕所有的f都是一邊偏向的(都大於0或都小於0),此時就有問題了,叫做定向多效性directional pleiotropy,這也是為什麼我們最後要做漏斗圖的原因。
就是通過漏斗圖一看都是所有的工具變數都是呈漏斗分佈的,就說明沒有偏向,這個時候我們認為定向多效性都被沖掉了,不影響。
好,解釋了上面的一些術語之後,我們實操一波。
實操
最基本的例子:BMI on CHD的例子,我想看一下BMI作為暴露,CHD作為結局的mr,程式碼就4條:
bmi_exp_dat <- extract_instruments(outcomes = 'ieu-a-2')
chd_out_dat <- extract_outcome_data(snps = bmi_exp_dat$SNP, outcomes = 'ieu-a-7')
dat <- harmonise_data(bmi_exp_dat, chd_out_dat)
res <- mr(dat)結果如下,下圖中有不同方法出來的我們關心的小b:
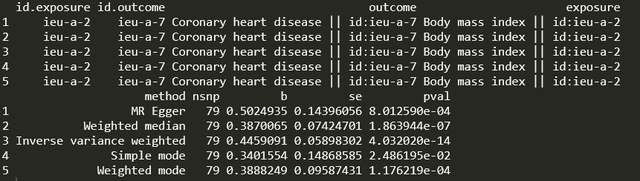
這個就算做完了,就這麼簡單快速。
接下來就是敏感性分析,首先是各個工具變數的異質性檢驗:
mr_heterogeneity(dat)執行程式碼後可以得到Cochran’s Q統計量
然後是水平基因多效性檢驗,程式碼如下:
mr_pleiotropy_test(dat)執行程式碼可以得到egger_intercept
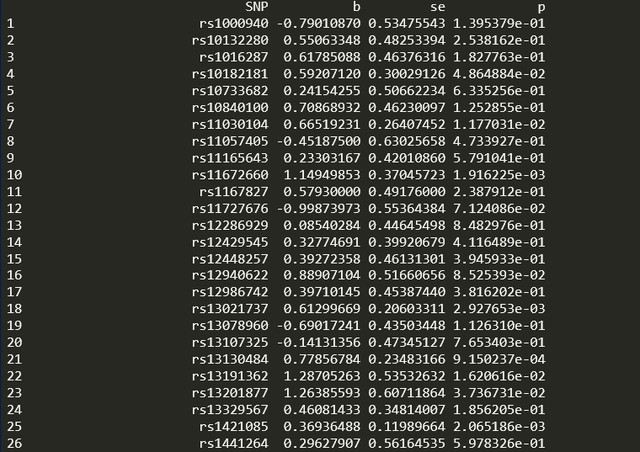
然後是單個SNP結果檢驗,程式碼如下:
res_single <- mr_singlesnp(dat)執行後可以得到每個SNP的小b
然後是留一檢驗,程式碼如下:
mr_leaveoneout(dat)接下來,論文中還會有幾個圖,首先是點圖,程式碼如下:
mr_scatter_plot(res, dat)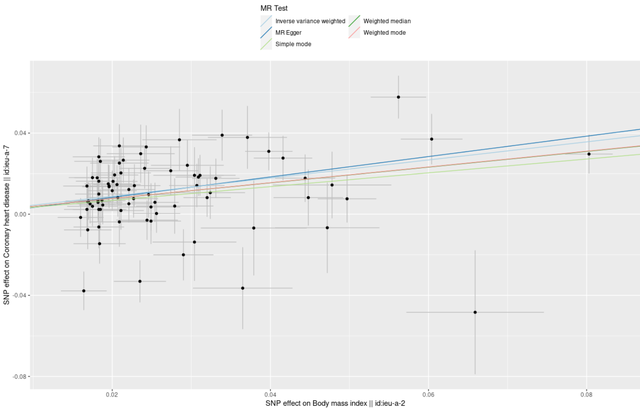
點圖是將同一個SNP對暴露的效果放在橫軸,對結局的效果放在縱軸,此時圖中的斜率就是我們的估計的小b。
然後是單個SNP效應組合的森林圖用mr_forest_plot函數可以得到,mr_leaveoneout_plot可以得到留一分析的森林圖,mr_funnel_plot可以幫我們得到漏斗圖。
到這就出了所有需要報告的東西,做完了。
但是上面的流程有很多的前提,比如你得知道暴露和結局的GWASid才能進行下去,GWAS又有很多,比如你直接用上面的程式碼的話其實是MR Base GWAS catalog裡面的GWAS,當然你還可以選別的,或者用自己找來的最新的GWAS都是可以的。

第一步首先是在相應的GWAS中找到暴露的summary data:
那麼有那些GWAS可以供我們使用呢?我們可以直接把GWAS的目錄調出來瞅瞅,程式碼如下:
data(gwas_catalog)執行後大約可以得到15萬個全基因組關聯研究的資料,截圖如下:
那麼對我們而言,我們現在需要找到我們關心的暴露對應的GWAS,比如我現在要找與「blood」表型相關的GWAS,我可以寫出如下程式碼:
exposure_gwas <- subset(gwas_catalog, grepl("Blood", Phenotype_simple))上面的程式碼相當於只用Phenotype_simple這一列做篩選,當然你也可以結合其它的列比如人群,比如作者,比如地區等等,都是可以的。
選好暴露相關的GWAS之後要做的就是進一步確定基因工具變數和暴露的強度,在論文中一般是這麼描述:First, relevance assumption was met considering that all SNPs have reached genome-wide significance (p < 5 × 10−8)
具體的操作如下:
exposure_gwas<-exposure_gwas[exposure_gwas$pval<5*10^-8,]通過上面的步驟保證我們的基因工具變數一定是和暴露強相關。
然後就是將準備好的暴露的GWAS資料形成可以用來做MR分析的資料格式,需要用到format_data()函數:
exposure_data<-format_data(exposure_gwas)此時的exposure_data大概長這樣:
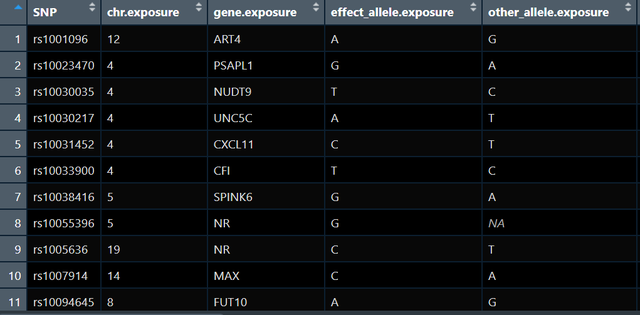
可以看到有很多個基因工具變數SNP,這個時候我們需要考慮連鎖不平衡(linkage disequilibrium):
exposure_data<-clump_data(exposure_data, clump_r2 = 0.001)上面的程式碼中clump_r2則是設定的容許相關性,到這兒我們算是手動地將工具變數都篩出來了,解決了找工具變數的問題,還有一個方法是自動篩選工具變數,比如我暴露是bmi,我可以寫出如下程式碼:
subset(ao, grepl("body mass", trait))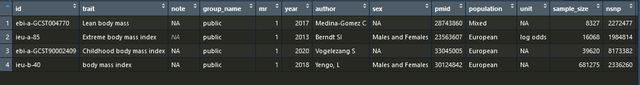
執行後我知道我可以選的gwasid是ieu-b-40,這個時候我也可以自動提取出工具變數,這兩種方法達到的目的都是一樣的:
extract_instruments('ieu-b-40')然後依照工具變數進行結局的summary estimates的提取,提取結局的summary data也需要是需要知道GWASid的對吧,比如我現在關心的結局是收縮壓,我就可以寫出如下程式碼:
outcome_gwas <- subset(ao, grepl("Systolic", trait))執行後我就可以知道所有的和收縮壓相關的gwasid了,我選一個最新的,比如我就選下面的2021年的:
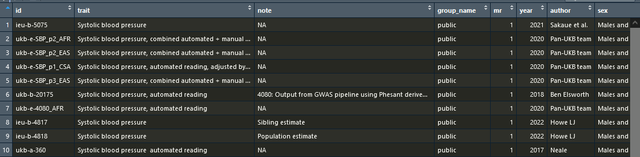
看圖我們知道它對於的id是ieu-b-5075,我就這麼寫:
outcome_data <- extract_outcome_data(
snps = exposure_data$SNP, outcomes = "ieu-b-5075")後續通過合併直接做mr分析就可以,流程就沒有不同了。
小結
今天給大家寫了孟德爾隨機話的實操,文章圖範例來自【中文孟德爾隨機化】英國布里斯托大學MRC-IEU《R語言做孟德爾隨機化》第一章:用MRBase網頁工具和R包TwoSampleMR做兩樣本孟德爾隨機化_嗶哩嗶哩_bilibili,感謝大家耐心看完