幾種常見取樣方法及原理
不平衡資料集是指類別分佈嚴重偏斜的資料集,例如少數類與多數類的樣本比例為 1:100 或 1:1000。
訓練集中的這種偏差會影響許多機器學習演演算法,甚至導致完全忽略少數類,容易導致模型過擬合,泛化能力差。
所以,針對類別分佈不均衡的資料集,一般會採取取樣的方式,使得類別分佈相對均衡,提升模型泛化能力。
下面介紹幾種常見的取樣方法及其原理,均是基於imbalanced-learn的實現:
1、樸素隨機取樣
隨機取樣:
- 隨機過取樣:從少數類中隨機選擇範例,並進行替換,然後將它們新增到訓練資料集中;
- 隨機欠取樣:從多數類中隨機選擇範例,並將它們從訓練資料集中刪除;
樸素重取樣,對資料沒有任何假設,也沒有使用啟發式方法。所以,易於實現且執行速度快,這對於非常大和複雜的資料集來說是ok的。
需注意的是,對類分佈的更改僅適用於訓練資料集,目的是優化模型的擬合;重取樣不適用於評估模型效能的測試集。
2、隨機過取樣
這種技術對於受偏態分佈影響並且給定類的多個重複範例會影響模型擬合的機器學習演演算法非常有效。
這可能包括迭代學習係數的演演算法,例如使用隨機梯度下降的人工神經網路,它還可能影響尋求資料良好拆分的模型,例如支援向量機和決策樹。
調整目標類分佈可能很有用,但在某些情況下,為嚴重不平衡的資料集尋求平衡分佈(因為它會精確複製少數類範例),可能會導致受影響的演演算法過度擬合少數類,從而導致泛化誤差增加。最好在過取樣後監控訓練和測試資料集的效能,並將結果與原始資料集上的相同演演算法進行比較。
# define oversampling strategy
oversample = RandomOverSampler(sampling_strategy='minority')
指定一個浮點值,指定轉換資料集中少數類與多數類範例的比率。比如,對於二元分類問題,假設對少數類進行過取樣,以使大多數類的範例數量減少一半,如果多數類有 1,000 個範例,而少數類有 100 個,則轉換後的資料集將有 500 個少數類的範例。
# define oversampling strategy
oversample = RandomOverSampler(sampling_strategy=0.5)
# fit and apply the transform
X_over, y_over = oversample.fit_resample(X, y)
完整範例:使用3個重複的 10 折交叉驗證進行評估,並在每1折內分別對訓練資料集執行過取樣
# example of evaluating a decision tree with random oversamplingfrom numpy import mean
from sklearn.datasets import make_classification
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.tree import DecisionTreeClassifier
from imblearn.pipeline import Pipeline
from imblearn.over_sampling import RandomOverSampler
# define dataset
X, y = make_classification(n_samples=10000, weights=[0.99], flip_y=0)
# define pipeline
steps = [('over', RandomOverSampler()), ('model', DecisionTreeClassifier())]
pipeline = Pipeline(steps=steps)
# evaluate pipeline
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3, random_state=1)
scores = cross_val_score(pipeline, X, y, scoring='f1_micro', cv=cv, n_jobs=-1)
score = mean(scores)
print('F1 Score: %.3f' % score)
3、隨機欠取樣
隨機欠取樣涉及從多數類中隨機選擇範例,並從訓練集中刪除。
欠取樣的一個限制是,刪除多數類中可能有用、重要或可能對擬合穩健決策邊界至關重要的範例(一不小心把重要資料刪了~~)。
鑑於範例是隨機刪除的,因此無法從多數類中檢測或保留好的或包含更多資訊的範例。資料的丟失會使少數和多數範例之間的決策邊界更難學習,從而導致分類效能下降。
# define undersample strategy
undersample = RandomUnderSampler(sampling_strategy='majority')
..
# define undersample strategy
undersample = RandomUnderSampler(sampling_strategy=0.5)
# fit and apply the transform
X_over, y_over = undersample.fit_resample(X, y)
4、隨機過取樣與欠取樣的結合
可以對少數類採用適度的過取樣以改善對這些範例的偏差,同時也可以對多數類採用適度的欠取樣以減少對該類的偏差。與單獨執行一種取樣相比,這可以提高模型整體效能。
例如,我們有一個類別分佈為 1:100 的資料集,可能首先應用過取樣,通過複製少數類的範例來將比例提高到 1:10,然後應用欠取樣,通過從多數類中刪除範例,將比例進一步提高到 1:2。
# define pipeline
over = RandomOverSampler(sampling_strategy=0.1)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('o', over), ('u', under), ('m', DecisionTreeClassifier())]
pipeline = Pipeline(steps=steps)
# example of evaluating a model with random oversampling and undersampling
from numpy import mean
from sklearn.datasets import make_classification
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.tree import DecisionTreeClassifier
from imblearn.pipeline import Pipeline
from imblearn.over_sampling import RandomOverSampler
from imblearn.under_sampling import RandomUnderSampler
# define dataset
X, y = make_classification(n_samples=10000, weights=[0.99], flip_y=0)
# define pipeline
over = RandomOverSampler(sampling_strategy=0.1)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('o', over), ('u', under), ('m', DecisionTreeClassifier())]
pipeline = Pipeline(steps=steps)
# evaluate pipeline
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3, random_state=1)
scores = cross_val_score(pipeline, X, y, scoring='f1_micro', cv=cv, n_jobs=-1)
score = mean(scores)
print('F1 Score: %.3f' % score)
5、其他幾類過取樣
5.1、SMOTE
RandomOverSampler通過複製少數類的一些原始樣本進行過取樣,而SMOTE 通過插值生成新樣本。
從現有範例中合成新範例,這是少數類資料增強的一種型別,被稱為合成少數類過取樣技術,簡稱SMOTE。
SMOTE 的工作原理是選擇特徵空間中接近的範例,在特徵空間中的範例之間繪製一條線,並在該線的某個點處繪製一個新樣本。
- SMOTE 首先隨機選擇一個少數類範例 a 並找到它的 k 個最近的少數類鄰居
- 然後通過隨機選擇 k 個最近鄰 b 中的一個並連線 a 和 b ,以在特徵空間中形成線段來建立合成範例,合成範例是作為兩個選定範例 a 和 b 的凸組合生成的。
建議首先使用隨機欠取樣來修剪多數類中的範例數量,然後使用 SMOTE 對少數類進行過取樣以平衡類分佈。SMOTE 和欠取樣的組合比普通欠取樣表現更好。(關於 SMOTE 的原始論文建議將 SMOTE 與多數類的隨機欠取樣結合起來)
普遍缺點在於,建立合成範例時沒有考慮多數類,如果類有很強的重疊,可能會導致範例不明確。
# Oversample and plot imbalanced dataset with SMOTEfrom collections import Counter
from sklearn.datasets import make_classification
from imblearn.over_sampling import SMOTE
from matplotlib import pyplot
from numpy import where
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# summarize class distribution
counter = Counter(y)
print(counter)
# transform the dataset
oversample = SMOTE()
X, y = oversample.fit_resample(X, y)
# summarize the new class distribution
counter = Counter(y)
print(counter)
# scatter plot of examples by class label
for label, _ in counter.items():
row_ix = where(y == label)[0]
pyplot.scatter(X[row_ix, 0], X[row_ix, 1], label=str(label))
pyplot.legend()
pyplot.show()
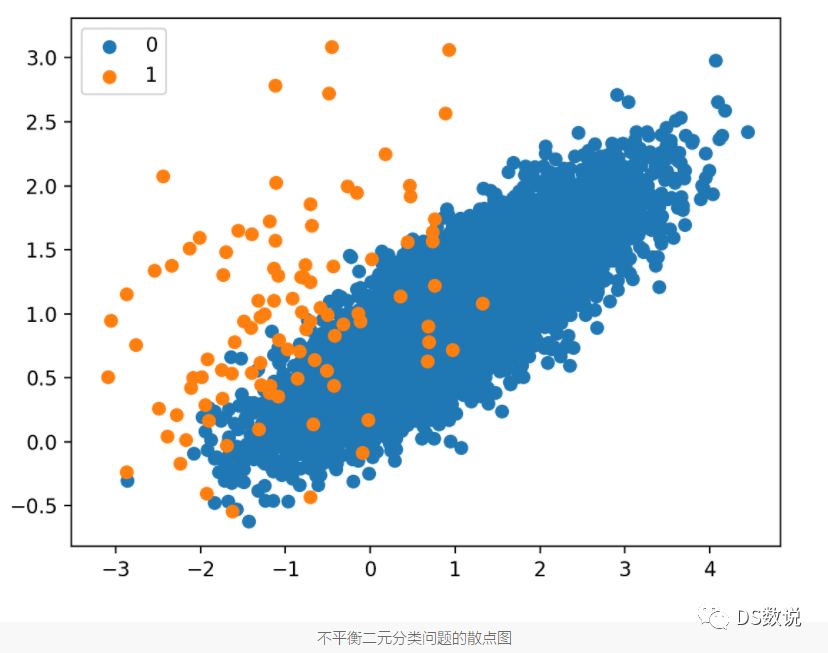
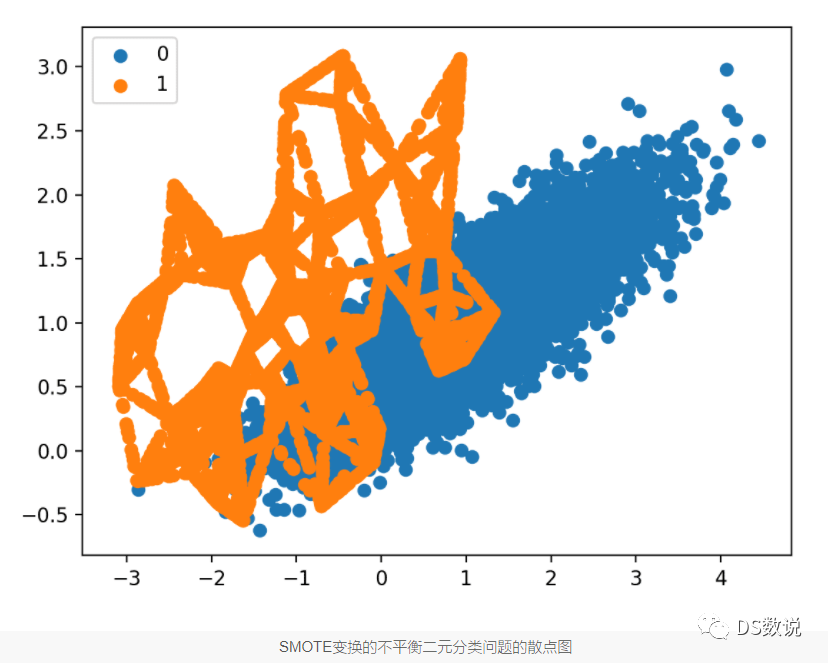
先對少數類smote過取樣,再樣對多數類欠取樣:
# Oversample with SMOTE and random undersample for imbalanced datasetfrom collections import Counter
from sklearn.datasets import make_classification
from imblearn.over_sampling import SMOTE
from imblearn.under_sampling import RandomUnderSampler
from imblearn.pipeline import Pipeline
from matplotlib import pyplot
from numpy import where
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# summarize class distribution
counter = Counter(y)
print(counter)
# define pipeline
over = SMOTE(sampling_strategy=0.1)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('o', over), ('u', under)]
pipeline = Pipeline(steps=steps)
# transform the dataset
X, y = pipeline.fit_resample(X, y)
# summarize the new class distribution
counter = Counter(y)
print(counter)
# scatter plot of examples by class label
for label, _ in counter.items():
row_ix = where(y == label)[0]
pyplot.scatter(X[row_ix, 0], X[row_ix, 1], label=str(label))
pyplot.legend()
pyplot.show()

可以對比取樣前和取樣後的模型效能(這裡取樣AUC):
# decision tree on imbalanced dataset with SMOTE oversampling and random undersamplingfrom numpy import mean
from sklearn.datasets import make_classification
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.tree import DecisionTreeClassifier
from imblearn.pipeline import Pipeline
from imblearn.over_sampling import SMOTE
from imblearn.under_sampling import RandomUnderSampler
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# define pipeline
model = DecisionTreeClassifier()
over = SMOTE(sampling_strategy=0.1)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('over', over), ('under', under), ('model', model)]
pipeline = Pipeline(steps=steps)
# evaluate pipeline
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3, random_state=1)
scores = cross_val_score(pipeline, X, y, scoring='roc_auc', cv=cv, n_jobs=-1)
print('Mean ROC AUC: %.3f' % mean(scores))
還可以通過調整 SMOTE 的 k 最近鄰的不同值(預設是5):
# grid search k value for SMOTE oversampling for imbalanced classificationfrom numpy import mean
from sklearn.datasets import make_classification
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import RepeatedStratifiedKFold
from sklearn.tree import DecisionTreeClassifier
from imblearn.pipeline import Pipeline
from imblearn.over_sampling import SMOTE
from imblearn.under_sampling import RandomUnderSampler
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# values to evaluate
k_values = [1, 2, 3, 4, 5, 6, 7]
for k in k_values:
# define pipeline
model = DecisionTreeClassifier()
over = SMOTE(sampling_strategy=0.1, k_neighbors=k)
under = RandomUnderSampler(sampling_strategy=0.5)
steps = [('over', over), ('under', under), ('model', model)]
pipeline = Pipeline(steps=steps)
# evaluate pipeline
cv = RepeatedStratifiedKFold(n_splits=10, n_repeats=3, random_state=1)
scores = cross_val_score(pipeline, X, y, scoring='roc_auc', cv=cv, n_jobs=-1)
score = mean(scores)
print('> k=%d, Mean ROC AUC: %.3f' % (k, score))

5.2、Borderline-SMOTE
SMOTE過於隨機了,從少數類中隨機選擇一個樣本a,找到K近鄰後,再從近鄰中隨機選擇一個樣本b,連線樣本a,b,選擇ab直線上一點de作為過取樣點。這樣很容易生成錯誤類樣本,生成的樣本進入到多數類中去了。
直覺:邊界上的範例和附近的範例比遠離邊界的範例更容易被錯誤分類,因此對分類更重要。
這些錯誤分類的範例可能是模稜兩可的,並且位於決策邊界的邊緣或邊界區域中,類別成員可能重疊。因此,這種修改為 SMOTE 的方法稱為 Borderline-SMOTE。
Borderline-SMOTE 方法僅在兩個類之間的決策邊界上建立合成範例,而不是盲目地為少數類生成新的合成範例。
# borderline-SMOTE for imbalanced datasetfrom collections import Counter
from sklearn.datasets import make_classification
from imblearn.over_sampling import BorderlineSMOTE
from matplotlib import pyplot
from numpy import where
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# summarize class distribution
counter = Counter(y)
print(counter)
# transform the dataset
oversample = BorderlineSMOTE()
X, y = oversample.fit_resample(X, y)
# summarize the new class distribution
counter = Counter(y)
print(counter)
# scatter plot of examples by class label
for label, _ in counter.items():
row_ix = where(y == label)[0]
pyplot.scatter(X[row_ix, 0], X[row_ix, 1], label=str(label))
pyplot.legend()
pyplot.show()
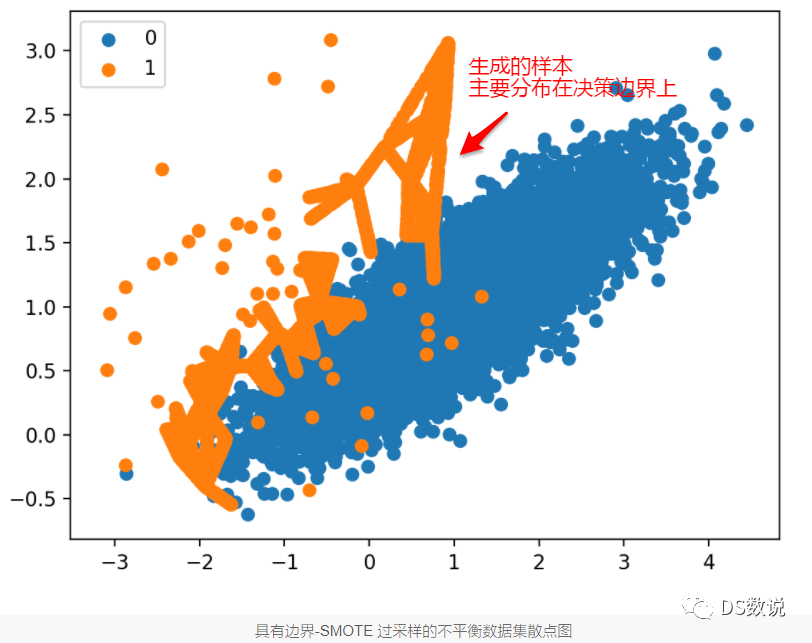
5.3 、Borderline-SMOTE SVM
與Borderline-SMOTE不同的是,使用 SVM 演演算法而不是 KNN 來識別決策邊界上的錯誤分類範例。
# borderline-SMOTE with SVM for imbalanced datasetfrom collections import Counter
from sklearn.datasets import make_classification
from imblearn.over_sampling import SVMSMOTE
from matplotlib import pyplot
from numpy import where
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# summarize class distribution
counter = Counter(y)
print(counter)
# transform the dataset
oversample = SVMSMOTE()
X, y = oversample.fit_resample(X, y)
# summarize the new class distribution
counter = Counter(y)
print(counter)
# scatter plot of examples by class label
for label, _ in counter.items():
row_ix = where(y == label)[0]
pyplot.scatter(X[row_ix, 0], X[row_ix, 1], label=str(label))
pyplot.legend()
pyplot.show()
與 Borderline-SMOTE 不同,更多的範例是在遠離類重疊區域的地方合成的,例如圖的左上角。
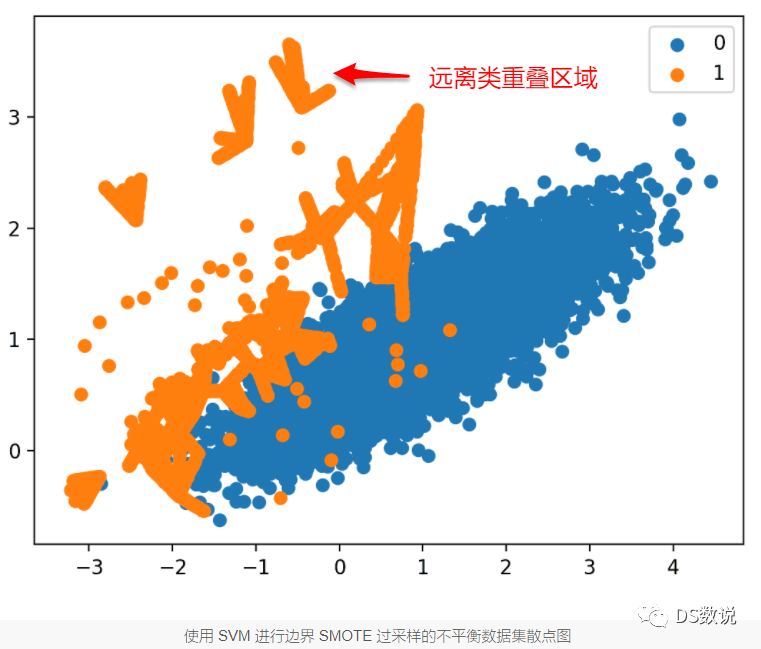
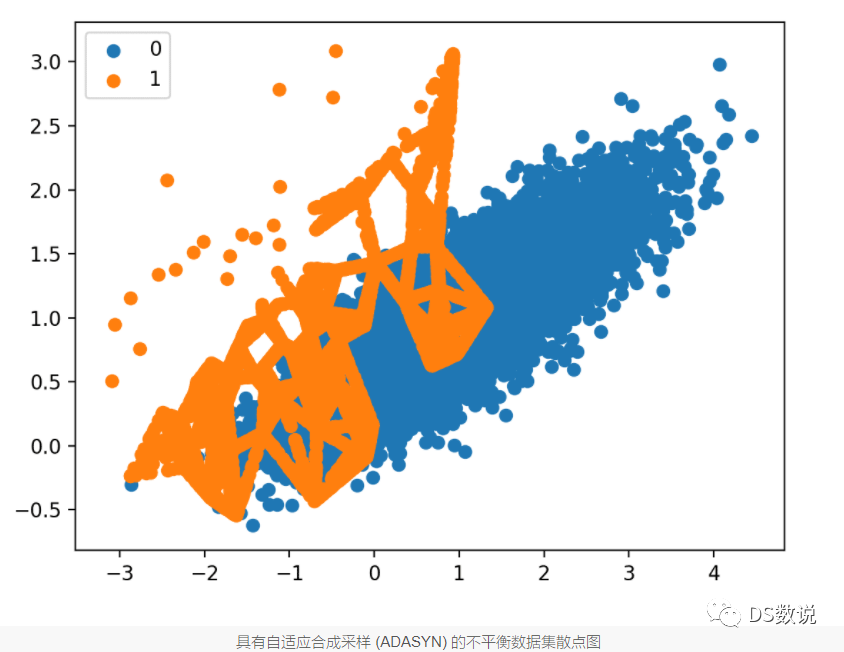
6、自適應合成取樣 (ADASYN)
通過生成與少數類中範例的密度成反比的合成樣本進行過取樣
也就是說,在特徵空間的少數樣本密度低的區域生成更多合成樣本,而在密度高的區域生成更少或不生成合成樣本。
ADASYN 演演算法的關鍵思想是,使用密度分佈作為標準,自動決定需要為每個少數資料範例生成的合成樣本的數量。
ADASYN 基於根據分佈自適應生成少數資料樣本的思想:與那些更容易學習的少數樣本相比,為更難學習的少數類樣本生成更多的合成資料。
# Oversample and plot imbalanced dataset with ADASYNfrom collections import Counter
from sklearn.datasets import make_classification
from imblearn.over_sampling import ADASYN
from matplotlib import pyplot
from numpy import where
# define dataset
X, y = make_classification(n_samples=10000, n_features=2, n_redundant=0,
n_clusters_per_class=1, weights=[0.99], flip_y=0, random_state=1)
# summarize class distribution
counter = Counter(y)
print(counter)
# transform the dataset
oversample = ADASYN()
X, y = oversample.fit_resample(X, y)
# summarize the new class distribution
counter = Counter(y)
print(counter)
# scatter plot of examples by class label
for label, _ in counter.items():
row_ix = where(y == label)[0]
pyplot.scatter(X[row_ix, 0], X[row_ix, 1], label=str(label))
pyplot.legend()
pyplot.show()
值得注意的一點是,在進行取樣的時候,要注意資料洩露問題。
在將整個資料集拆分為訓練分割區和測試分割區之前對整個資料集進行重取樣,這就會導致資料洩露。
-
該模型不會在類分佈類似於真實用例的資料集上進行測試。通過對整個資料集進行重新取樣,訓練集和測試集都可能是平衡的,但是模型應該在不平衡的資料集上進行測試,以評估模型的潛在偏差;
-
重取樣過程可能會使用有關資料集中樣本的資訊來生成或選擇一些樣本。因此,我們可能會使用樣本資訊,這些資訊將在以後用作測試樣本。
References:
- https://imbalanced-learn.org/dev/user_guide.html
- https://zhuanlan.zhihu.com/p/360045341
歡迎關注個人公眾號:DS數說