分散式機器學習:邏輯迴歸的並行化實現(PySpark)
1. 梯度計算式匯出
我們在部落格《統計學習:邏輯迴歸與交叉熵損失(Pytorch實現)》中提到,設\(w\)為權值(最後一維為偏置),樣本總數為\(N\),\(\{(x_i, y_i)\}_{i=1}^N\)為訓練樣本集。樣本維度為\(D\),\(x_i\in \mathbb{R}^{D+1}\)(最後一維擴充),\(y_i\in\{0, 1\}\)。則邏輯迴歸的損失函數為:
這裡
寫成這個形式就已經可以用諸如Pytorch這類工具來進行自動求導然後採用梯度下降法求解了。不過若需要用表示式直接計算出梯度,我們還需要將損失函數繼續化簡為:
可將梯度表示如下:
2. 基於Spark的並行化實現
邏輯迴歸的目標函數常採用梯度下降法求解,該演演算法的並行化可以採用如下的Map-Reduce架構:
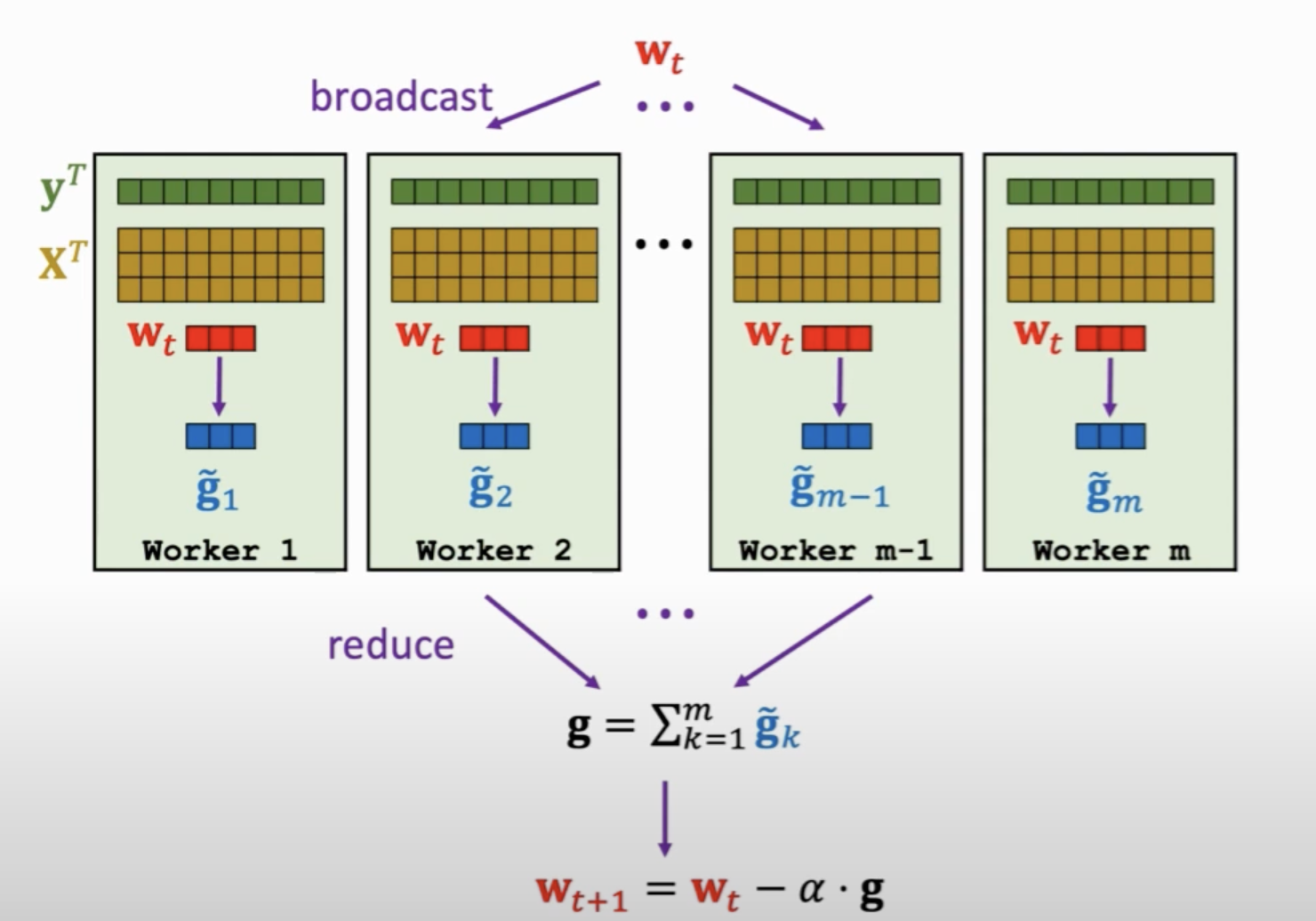
先將第\(t\)輪迭代的權重廣播到各worker,各worker計算一個區域性梯度(map過程),然後再將每個節點的梯度聚合(reduce過程),最終對引數進行更新。
在Spark中每個task對應一個分割區,決定了計算的並行度(分割區的概念詳間我們上一篇部落格《Spark: 單詞計數(Word Count)的MapReduce實現(Java/Python)》 )。在Spark的實現過程如下:
-
map階段: 各task執行
map()函數對每個樣本\((x_i, y_i)\)計算梯度\(g_i\), 然後對每個樣本對應的梯度執行進行本地聚合,以減少後面的資料傳輸量。如第1個task執行reduce()操作得到\(\widetilde{g}_1 = \sum_{i=1}^3 g_i\) 如下圖所示: -
reduce階段:使用
reduce()將所有task的計算結果收集到Driver端進行聚合,然後進行引數更新。
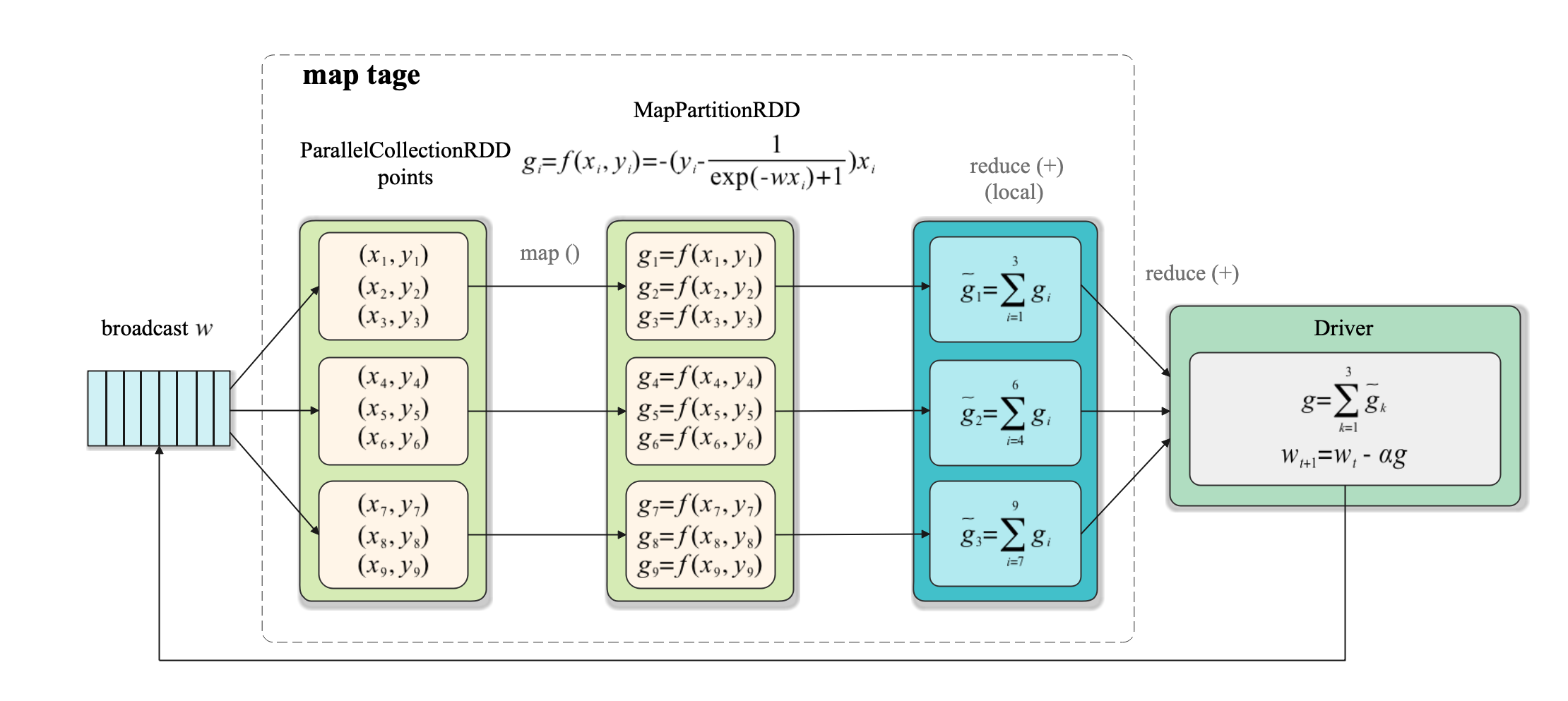
在上圖中,訓練資料用points:PrallelCollectionRDD來表示,引數向量用\(w\)來表示,注意引數向量不是RDD,只是一個單獨的參與運算的變數。
此外需要注意一點,雖然每個task在本地進行了區域性聚合,但如果task過多且每個task本地聚合後的結果(單個gradient)過大那麼統一傳遞到Driver端仍然會造成單點的網路平均等問題。為了解決這個問題,Spark設計了效能更好的treeAggregate()操作,使用樹形聚合方法來減少網路和計算延遲。
3. PySpark實現程式碼
PySpark的完整實現程式碼如下:
from sklearn.datasets import load_breast_cancer
import numpy as np
from pyspark.sql import SparkSession
from operator import add
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
n_slices = 3 # Number of Slices
n_iterations = 300 # Number of iterations
alpha = 0.01 # iteration step_size
def logistic_f(x, w):
return 1 / (np.exp(-x.dot(w)) + 1)
def gradient(point: np.ndarray, w: np.ndarray) -> np.ndarray:
""" Compute linear regression gradient for a matrix of data points
"""
y = point[-1] # point label
x = point[:-1] # point coordinate
# For each point (x, y), compute gradient function, then sum these up
return - (y - logistic_f(x, w)) * x
if __name__ == "__main__":
X, y = load_breast_cancer(return_X_y=True)
D = X.shape[1]
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.3, random_state=0)
n_train, n_test = X_train.shape[0], X_test.shape[0]
spark = SparkSession\
.builder\
.appName("Logistic Regression")\
.getOrCreate()
matrix = np.concatenate(
[X_train, np.ones((n_train, 1)), y_train.reshape(-1, 1)], axis=1)
points = spark.sparkContext.parallelize(matrix, n_slices).cache()
# Initialize w to a random value
w = 2 * np.random.ranf(size=D + 1) - 1
print("Initial w: " + str(w))
for t in range(n_iterations):
print("On iteration %d" % (t + 1))
g = points.map(lambda point: gradient(point, w)).reduce(add)
w -= alpha * g
y_pred = logistic_f(np.concatenate(
[X_test, np.ones((n_test, 1))], axis=1), w)
pred_label = np.where(y_pred < 0.5, 0, 1)
acc = accuracy_score(y_test, pred_label)
print("iterations: %d, accuracy: %f" % (t, acc))
print("Final w: %s " % w)
print("Final acc: %f" % acc)
spark.stop()
注意spark.sparkContext.parallelize(matrix, n_slices)中的n_slices就是Spark中的分割區數。我們在程式碼中採用breast cancer資料集進行訓練和測試,該資料集是個二分類資料集。模型初始權重採用隨機初始化。
最後,我們來看一下演演算法的輸出結果。
初始權重如下:
Initial w: [-0.0575882 0.79680833 0.96928013 0.98983501 -0.59487909 -0.23279241
-0.34157571 0.93084048 -0.10126002 0.19124314 0.7163746 -0.49597826
-0.50197367 0.81784642 0.96319482 0.06248513 -0.46138666 0.76500396
0.30422518 -0.21588114 -0.90260279 -0.07102884 -0.98577817 -0.09454256
0.07157487 0.9879555 0.36608845 -0.9740067 0.69620032 -0.97704433
-0.30932467]
最終的模型權重與在測試集上的準確率結果如下:
Final w: [ 8.22414803e+02 1.48384087e+03 4.97062125e+03 4.47845441e+03
7.71390166e+00 1.21510016e+00 -7.67338147e+00 -2.54147183e+00
1.55496346e+01 6.52930570e+00 2.02480712e+00 1.09860082e+02
-8.82480263e+00 -2.32991671e+03 1.61742379e+00 8.57741145e-01
1.30270454e-01 1.16399854e+00 2.09101988e+00 5.30845885e-02
8.28547658e+02 1.90597805e+03 4.93391021e+03 -4.69112527e+03
1.10030574e+01 1.49957834e+00 -1.02290791e+01 -3.11020744e+00
2.37012097e+01 5.97116694e+00 1.03680530e+02]
Final acc: 0.923977
可見我們的演演算法收斂良好。
參考
- [1] GiHub: Spark官方Python樣例
- [2] 王樹森-平行計算與機器學習(1/3)
- [3] 劉鐵巖,陳薇等. 分散式機器學習:演演算法、理論與時間[M]. 機械工業出版社, 2018.
- [4] 許利傑,方亞芬. 巨量資料處理框架Apache Spark設計與實現[M]. 電子工業出版社, 2021.